治疗下肿瘤生长动力学的PK/PD模型
本文提出了一个SimBiology模型,描述了抗癌药物的药代动力学/药效学(PK/PD)行为,如文中所述,应用抗癌药物后异种移植模型中肿瘤生长动力学的预测药代动力学建模由Simeoni等人。
模型描述
2004年,Simeoni等[1]提出了一个PK/PD模型来量化抗癌药物对肿瘤生长动力学的影响在活的有机体内动物研究。药物的药代动力学由一个2室模型描述,该模型有静脉给药和线性消除(柯)从中央隔间。肿瘤生长被描述为一个双阶段的过程,最初的指数增长,然后是线性增长。增殖肿瘤细胞的生长速度,x1,描述为:
在那里,
而且
是肿瘤生长参数,
x1增殖的肿瘤细胞的重量,和w为肿瘤总重量。在没有药物的情况下,肿瘤仅由增殖细胞组成。w = x1.在抗癌剂存在的情况下,假设一小部分增殖细胞转化为非增殖细胞。这种转化的速率被认为是血浆药物浓度和疗效因子的函数,k2.非增殖细胞,x2,经过一系列的转运阶段(x3而且x4),并最终从系统中清除。中转期的加入是为了将药物加入和肿瘤重量明显减轻之间的延迟纳入其中。通过中转舱的流动被建模为一阶过程(k1).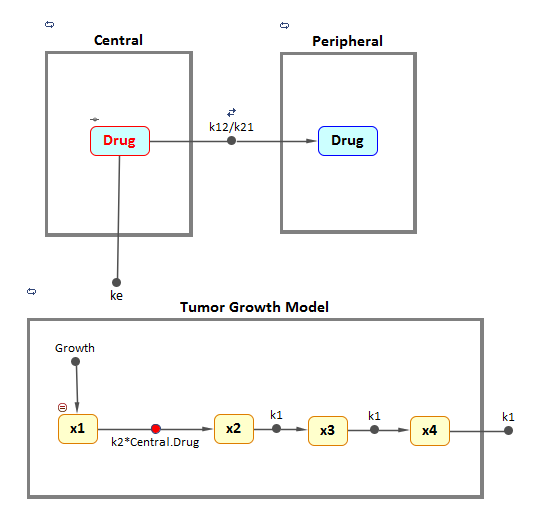
参考文献
[1] Simeoni M, Magni P, Cammia C, De Nicolao G, Croci V, Pesenti E, Germani M, Poggesi I和Rocchetti M(2004)应用抗癌药物后异种移植物模型中肿瘤生长动力学的预测药代动力学建模。癌症研究64:1094-1101
