识别重要特征和分类蛋白质谱
这个例子展示了如何对质谱数据进行分类,并使用一些统计工具来寻找潜在的疾病标志物和蛋白质组学模式诊断。
介绍
血清蛋白质组学诊断可用于区分有疾病和无疾病患者的样本。使用表面增强激光解吸和电离(SELDI)蛋白质谱生成谱图。这项技术有可能改善癌症病理的临床诊断测试。目标是选择一组可用于区分癌症患者和对照组患者的测量值或“特征”。这些特征将是特定质量/电荷值下的离子强度水平。
数据进行预处理
本例中的卵巢癌数据集来自FDA-NCI临床蛋白质组学程序数据库.数据集使用WCX2蛋白阵列生成。该数据集包括95名对照组和121名卵巢癌患者。有关该数据集的详细描述,请参见[1]和[4]。
本示例假设您已经拥有预处理的数据OvarianCancerQAQCdataset.mat.但是,如果没有数据文件,可以按照示例中的步骤重新创建使用顺序和并行计算的光谱批处理.
或者,您也可以运行该脚本msseqprocessing.m.
目录(fullfile (matlabroot,“例子”,“bioinfo”,“主要”))%确保支持文件在搜索路径上金宝app类型msseqprocessing
% MSSEQPROCESSING脚本创建OvarianCancerQAQCdataset。mat(用于% CANCERDETECTDEMO)。在运行此文件之前,将变量% "repository"初始化为您放置质谱%文件的完整路径。例如:% % repository = 'F:/MassSpecRepository/OvarianCD_PostQAQC/';% % or % % repository = '/home/username/MassSpecRepository/ ovanancd_postqaqc /';% %执行时间大约为18分钟(奔腾4,4ghz)。如果您有并行计算工具箱,请参考BIODISTCOMPDEMO,以了解如何加快此分析。% Copyright 2003-2008 The MathWorks, Inc. repositoryC = [repository 'Cancer/'];[存储库'Normal/'];filesCancer = dir([repositoryC '*.txt']);NumberCancerDatasets =元素个数(filesCancer); fprintf('Found %d Cancer mass-spectrograms.\n',NumberCancerDatasets) filesNormal = dir([repositoryN '*.txt']); NumberNormalDatasets = numel(filesNormal); fprintf('Found %d Control mass-spectrograms.\n',NumberNormalDatasets) files = [ strcat('Cancer/',{filesCancer.name}) ... strcat('Normal/',{filesNormal.name})]; N = numel(files); % total number of files fprintf('Total %d mass-spectrograms to process...\n',N) [MZ,Y] = msbatchprocessing(repository,files); disp('Finished; normalizing and saving to OvarianCancerQAQCdataset.mat.') Y = msnorm(MZ,Y,'QUANTILE',0.5,'LIMITS',[3500 11000],'MAX',50); grp = [repmat({'Cancer'},size(filesCancer));... repmat({'Normal'},size(filesNormal))]; save OvarianCancerQAQCdataset.mat Y MZ grp
上面列出的脚本和示例中的预处理步骤旨在说明一组具有代表性的可能的预处理过程。使用不同的步骤或参数可能会导致本示例的不同结果,甚至可能得到改进。
加载数据
一旦有了预处理的数据,就可以将其加载到MATLAB中。
负载OvarianCancerQAQCdataset谁
名称大小字节类属性MZ 15000x1 120000 double Y 15000x216 25920000 double grp 216x1 25056 cell
有三个变量:MZ,Y,grp.MZ是质量/电荷矢量,Y是否为所有216例患者(对照组和癌症患者)的强度值grp保存这些样本中哪些代表癌症患者,哪些代表正常患者的索引信息。
初始化将在整个示例中使用的一些变量。
N =元素个数(grp);%样本数量Cidx = strcmp (“癌症”、grp);%癌症样本的逻辑索引向量Nidx = strcmp (“正常”、grp);%用于正常样本的逻辑索引向量Cvec =找到(Cidx);%癌症样本索引向量Nvec =找到(Nidx);%指标向量为正常样本xAxisLabel =“质量/电荷(M / Z)”;% x标号用于绘图yAxisLabel =离子强度的;图的% y标签
可视化一些示例
您可以将一些数据集绘制到一个图形窗口中,以直观地比较两组的概况;在这个例子中显示了5个癌症患者的光谱图(蓝色)和5个对照患者的光谱图(绿色)。
图;持有在;hC =情节(MZ, Y (:, Cvec (1:5)),“b”);hN =情节(MZ, Y (:, Nvec (1:5)),‘g’);包含(xAxisLabel);ylabel (yAxisLabel);[hN(1),hC(1)],{“控制”,卵巢癌的})标题(多个样品谱图的)

将区域从8500 M/Z放大到8700 M/Z,可以看到一些可能对数据分类有用的峰值。
轴([8450、8700、1、7])
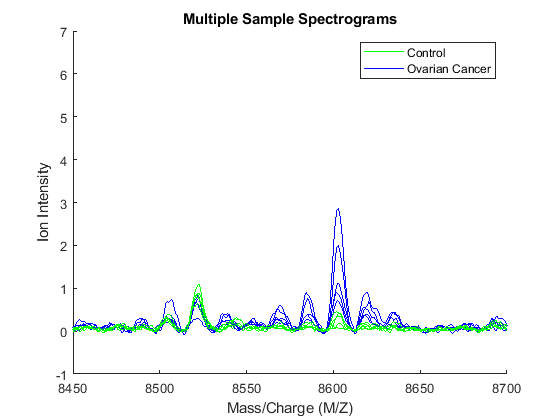
另一种可视化整个数据集的方法是查看对照组和癌症样本的组平均信号。你可以画出组平均值和每个组的信封。
mean_N =意味着(Y (:, Nidx), 2);%组平均值为对照样品max_N = max (Y (:, Nidx), [], 2);%顶部信封的对照样品min_N = min (Y (:, Nidx), [], 2);%底部信封的对照样品mean_C =意味着(Y (:, Cidx), 2);%组平均癌症样本max_C = max (Y (:, Cidx), [], 2);%顶部信封的对照样品min_C = min (Y (:, Cidx), [], 2);%底部信封的对照样品图;持有在;hC =情节(MZ mean_C,“b”);hN =情节(MZ mean_N,‘g’);MZ,[max_C min_C],“b——”);n = MZ,[max_N min_N],“g——”);包含(xAxisLabel);ylabel (yAxisLabel);轴([8450、8700、1、7])传说([hN, hC, gN (1) gC (1)), {对照组Avg。,“卵巢癌组平均值”,...“控制信封”,卵巢癌信封的},...“位置”,“西北”)标题(“组别平均数字及组别信封”)

观察一下,显然没有一个单一的特征可以完美地区分两个群体。
排名关键特性
寻找重要特性的一个简单方法是假设每个M/Z值是独立的,并计算一个双向t检验。rankfeatures返回最重要的M/Z值的索引,例如按测试统计数据的绝对值排列的100个索引。这种特征选择方法也被称为过滤方法,其中学习算法不涉及如何选择特征。
[壮举,统计]= rankfeatures (Y, grp,“标准”,的tt,“数量”, 100);
的第一个输出rankfeatures可用于提取重要特征的M/Z值。
sig_Masses = MZ(成绩);sig_Masses (1:7)%显示前7个
Ans = 1.0e+03 * 8.1009 8.1016 8.1024 8.1001 8.1032 7.7366 7.7359
的第二个输出rankfeatures为检验统计量绝对值的向量。你可以用yyaxis.
图;yyaxis左情节(MZ (mean_N mean_C]);ylim ([20]) xlim((7950、8300))标题(“重要的M / Z值”)包含(xAxisLabel);ylabel (yAxisLabel);yyaxis正确的情节(MZ,统计);ylim ([22]) ylabel (检验统计量的);传奇({对照组Avg。,“卵巢癌组织平均值”,“测试数据”})

注意,在高M/Z值但低强度(~8100 Da.)有显著区域。中还有其他度量类可分离性的方法rankfeatures,如基于熵的,Bhattacharyya,或经验接受者工作特征(ROC)曲线下的面积。
基于线性判别分析的盲分类
现在您已经确定了一些重要的特征,您可以使用这些信息对癌症和正常样本进行分类。由于样本数量较少,您可以使用20%坚持值运行交叉验证,以更好地估计分类器的性能。cvpartition可设置hold out、K-fold、Leave-M-Out等不同类型系统评价方法的培训和测试指标。
per_eval = 0.20;交叉验证培训规模%rng (“默认”);初始化随机生成器到相同的状态%用于生成已发布的示例简历= cvpartition (grp),“坚持”per_eval)
nummobations: 216 NumTestSets: 1 TrainSize: 173 TestSize: 43
观察特征仅从训练子集中选择,并使用测试子集执行验证。classperf允许您跟踪多个验证。
cp_lda1 = classperf (grp);初始化CP对象为k = 1:10进行交叉验证10次简历=重新分区(简历);壮举= rankfeatures (Y(:,培训(cv)、grp(培训(cv)),“数量”, 100);c =分类(Y(功绩,测试(cv)), Y(成绩、培训(cv)), grp(培训(cv)));classperf (cp_lda1 c测试(cv));%使用当前验证更新CP对象结束
在循环之后,您可以使用CP对象中的任何属性(如错误率、敏感性、特异性等)来评估总体盲分类的性能。
cp_lda1
标签:“描述:“ClassLabels: {2 x1细胞}GroundTruth: x1双[216]NumberOfObservations: 216 ControlClasses: 2 TargetClasses: 1 ValidationCounter: 10 SampleDistribution: x1双[216]ErrorDistribution: x1双[216]SampleDistributionByClass: [2 x1双]ErrorDistributionByClass: [2 x1双]CountingMatrix:[3x2 double] CorrectRate: 0.8488 ErrorRate: 0.1512 LastCorrectRate: 0.8837 LastErrorRate: 0.1163 InconclusiveRate: 0 ClassifiedRate: 1敏感性:0.8208特异性:0.8842 positive predictivvalue: 0.8995 negative predictivvalue: 0.7962 positive可能性:7.0890 negative可能性:0.2026发病率:0.5581诊断表:[2x2 double]
这种单纯的特征选择方法可以通过基于区域信息去除一些特征来改进。例如,'NWEIGHT'rankfeatures超过相邻M/Z特征的检验统计量,从而可以将其他显著的M/Z值合并到选定特征的子集中
cp_lda2 = classperf (grp);初始化CP对象为k = 1:10进行交叉验证10次简历=重新分区(简历);壮举= rankfeatures (Y(:,培训(cv)、grp(培训(cv)),“数量”, 100,“NWEIGHT”5);c =分类(Y(功绩,测试(cv)), Y(成绩、培训(cv)), grp(培训(cv)));classperf (cp_lda2 c测试(cv));%使用当前验证更新CP对象结束cp_lda2。CorrectRate%平均正确分类率
ans = 0.9023
数据维数的PCA/LDA降维
Lilien等人在[2]中提出了一种利用主成分分析(PCA)降低数据维数的算法,然后使用LDA对组进行分类。在这个例子中,M/Z空间中2000个最重要的特征被映射到150个主分量
cp_pcalda = classperf (grp);初始化CP对象为k = 1:10进行交叉验证10次简历=重新分区(简历);%选择2000个最重要的特征。壮举= rankfeatures (Y(:,培训(cv)、grp(培训(cv)),“数量”, 2000);% PCA降维P = pca (Y(成绩、培训(cv)));%项目到PCA空间x = Y(feat,:)' * P(:, 1:30 0);%利用LDAc =分类(x(测试(cv):), x(培训(cv):), grp(培训(cv)));classperf (cp_pcalda c测试(cv));结束cp_pcalda。CorrectRate%平均正确分类率
ans = 0.9814
子集特征选择的随机搜索
特征选择也可以通过分类来加强,这种方法通常被称为包装器选择方法。特征选择的随机搜索生成特征的随机子集,并使用学习算法独立地评估它们的质量。之后,它会选择最常见的好特征。Li等人在[3]中将这一概念应用于蛋白质表达模式的分析。的randfeatures函数允许您使用LDA或在随机特征子集上使用k-最近邻分类器搜索特征子集。
注意:下面的示例是计算密集型的,因此在示例中禁用了它。此外,为了获得更好的结果,您应该从中的默认值增加池大小和分类器的严格程度randfeatures.类型帮助randfeatures为更多的信息。
如果0% <==更改为1以启用。这可能需要大量的时间来完成。简历=重新分区(简历);[壮举,fCount] = randfeatures (Y(:,培训(cv)、grp(培训(cv)),...“分类”,“哒”,“PerformanceThreshold”, 0.90);其他的负载randFeatCancerDetect结束
使用评估集评估选定特征的质量
的第一个输出randfeatures是MZ值索引的有序列表。第一项最常出现在获得良好分类的子集中。第二个输出是每个值被选择的实际次数。您可以使用嘘来看看这个分布。
图;嘘(fCount,马克斯(fCount) + 1);
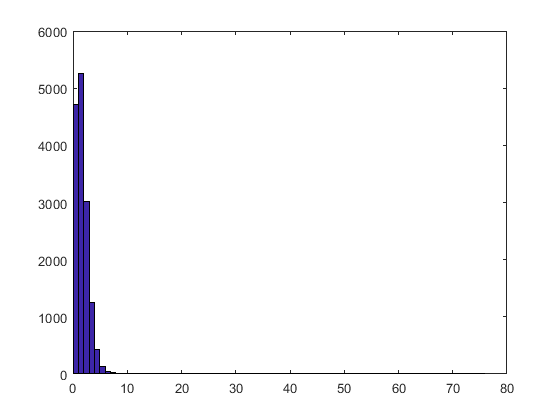
您将看到,在选定的子集中,大多数值最多只出现一次。放大可以更好地了解更频繁选择的值的细节。
轴([0 80 0 100])

只有少数几个值被选择了10次以上。您可以通过使用茎图来显示最常选择的特征来可视化这些特征。
图;持有在;sigFeats = fCount;sigFeats (sigFeats < = 10) = 0;情节(MZ (mean_N mean_C]);茎(MZ (sigFeats > 0) sigFeats (sigFeats > 0),“r”);轴([2000、12000、80])传说({对照组Avg。,“卵巢癌组织平均值”,的显著特征},...“位置”,“西北”)包含(xAxisLabel);ylabel (yAxisLabel);

这些特征似乎聚集成几组。通过运行下面的实验,您可以进一步研究有多少特性是重要的。首先选取最频繁的特征对数据进行分类,然后选取最频繁的两个特征,以此类推,直到选取10次以上的特征全部被使用。然后,您可以看到添加更多特性是否会改进分类器。
nSig =总和(fCount > 10);nSig cp_rndfeat = 0(20日);为我= 1:nSig为J = 1:20 CV =重分区;P = pca (Y(壮举(1:我)、培训(cv)) ");x = Y(feat(1:i),:)' * P;c =分类(x(测试(cv):), x(培训(cv):), grp(培训(cv)));cp = classperf (grp c测试(cv));cp_rndfeat (j, i) = cp.CorrectRate;%平均正确分类率结束结束图的阴谋(1:nSig,[马克斯(cp_rndfeat);意味着(cp_rndfeat)]);传奇({“最佳CorrectRate”,“意思是CorrectRate”},“位置”,“东南”)

从这张图中你可以看到,即使只有三个特征,有时也有可能得到完美的分类。你还会注意到,平均正确率的最大值出现在少数特征上,然后逐渐下降。
[bestAverageCR, bestNumFeatures] = max(mean(cp_rndfeat));
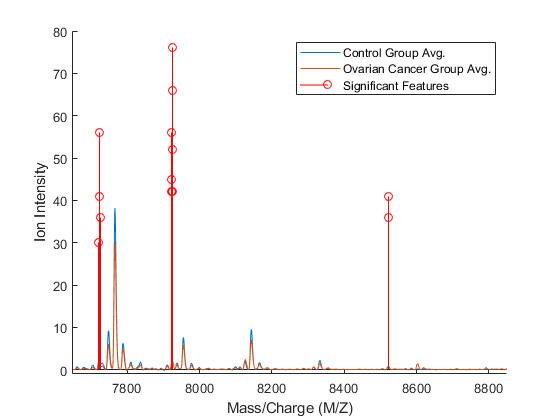
现在,您可以可视化提供最佳平均分类的特性。你可以看到这些实际上只对应数据中的三个峰值。
图;持有在;sigFeats = fCount;sigFeats (sigFeats < = 10) = 0;ax_handle = plot(MZ,[mean_N mean_C]);茎(MZ(壮举(1:bestNumFeatures)), sigFeats(壮举(1:bestNumFeatures)),“r”);轴([7650、8850、80])传说({对照组Avg。,“卵巢癌组织平均值”,的显著特征})包含(xAxisLabel);ylabel (yAxisLabel);
备选统计学习算法
在MATLAB®中有许多分类工具,您也可以使用它们来分析蛋白质组数据。其中有支持向量机(金宝appfitcsvm), k近邻(fitcknn)、神经网络(深度学习工具箱™)和分类树(fitctree).对于特征选择,您还可以使用顺序子集特征选择(sequentialfs)或使用遗传算法(全局优化工具箱)优化随机搜索方法。例如,请参见质谱数据特征的遗传算法搜索.
参考文献
[1]Conrads, T P, V A Fusaro, S Ross, D Johann, V Rajapakse, B A Hitt, S M Steinberg,等“高分辨率血清蛋白质组学特征用于卵巢癌检测”。内分泌相关癌症,2004年6月,163-78。
[2]莉莉安,瑞安·H,哈尼·法里德,布鲁斯·r·唐纳德。人血清质谱表达依赖蛋白组学数据的概率疾病分类《计算生物学杂志》,第10期。6(2003年12月):925-46。
[3]李,L., D. M. Umbach, P. Terry,和J. A. Taylor。遗传算法/KNN方法在SELDI蛋白质组学数据中的应用生物信息学20,不。10(2004年7月1日):1638-40。
[4]Petricoin, Emanuel F, Ali M Ardekani, Ben A Hitt, Peter J Levine, Vincent A Fusaro, Seth M Steinberg, Gordon B Mills,等。《柳叶刀》359号,no。9306(2002年二月):572-77。

