变异变异和交叉
设置突变的数量
遗传算法使用MutationFcn选择。默认的突变选项,@mutationgaussian,加一个随机数,或突变,从高斯分布中选择,到父向量的每个项。通常情况下,突变的数量,与分布的标准差成正比,在每一代新的时候都会减少。你可以控制算法在每一代中应用到父代的平均变异量规模和缩小单元格数组中包含的输入:
选择= optimoptions (“遗传算法”,...“MutationFcn”, {@mutationgaussian规模缩小});
规模和缩小标量有默认值吗1每一个。
规模控制第一代突变的标准差。这个值是规模乘以初始总体的范围,也就是InitialPopulationRange选择。缩小控制平均变异量减少的速度。标准差线性减小,最终值为1 -缩小乘以第一代的初始值。例如,如果缩小默认值为1,则突变量在最后一步降到0。
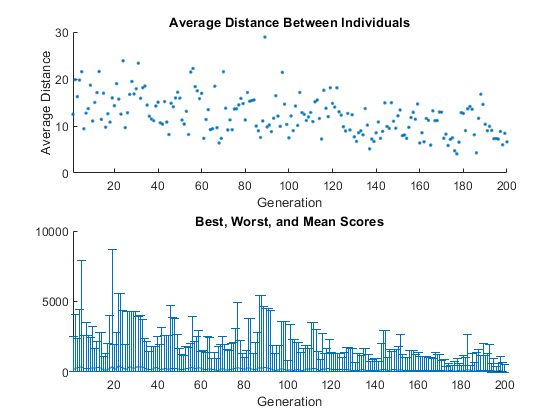
通过选择plot函数,可以看到突变的效果@gaplotdistance和@gaplotrange,然后在一个问题上运行遗传算法,如在最小化Rastrigin的功能.下图显示了设置随机数生成器后的图。
rng默认的%的再现性选择= optimoptions (“遗传算法”,“PlotFcn”{@gaplotdistance, @gaplotrange},...“MaxStallGenerations”, 200);才能跑得更远[x, fval] = ga (@rastriginsfcn 2 ,[],[],[],[],[],[],[], 选项);

上面的图显示了每一代点之间的平均距离。随着突变数量的减少,个体之间的平均距离也在减少,在最后一代时,平均距离大约为0。下面的图显示了每一代的一条垂线,显示了从最小到最大的适应度值以及平均适应度值的范围。随着变异数量的减少,变异范围也在减少。这些图表明,减少突变量会降低后代的多样性。
为便于比较,下图显示了您设置时相同的图缩小来0.5.
选择= optimoptions (“遗传算法”选项,...“MutationFcn”, {@mutationgaussian 1。5});[x, fval] = ga (@rastriginsfcn 2 ,[],[],[],[],[],[],[], 选项);
这一次,变异的平均数量在最后一代中减少了1/2。因此,个体之间的平均距离比以前减少得更少。
设置交叉分数
的CrossoverFraction选项指定除精英儿童外,由跨界儿童组成的每个人口的比例。的交叉分数1意味着除精英个体外的所有孩子都是跨界儿童,而跨界比例为0意思是所有的孩子都是变异的孩子。下面的例子表明,这两种极端都不是优化函数的有效策略。
该示例使用适应度函数,该函数在某一点的值是各点坐标绝对值的和。也就是说,
通过将适应度函数设置为,可以将此函数定义为匿名函数
@ (x)和(abs (x))
运行默认值为的示例0.8随着CrossoverFraction选择。
Fun = @(x) sum(abs(x));据nvar = 10;选择= optimoptions (“遗传算法”,...“InitialPopulationRange”(1, 1),...“PlotFcn”, {@gaplotbestf, @gaplotdistance});rng(14日“旋风”)%的再现性[x, fval] = ga(有趣,据nvar ,[],[],[],[],[],[],[], 选项)

优化终止:适应度值的平均变化小于选项。X = -0.0020 -0.0134 -0.0067 -0.0028 -0.0241 -0.0118 0.0021 0.0113 -0.0021 -0.0036 fval = 0.0799
交叉,变异
要查看没有突变时遗传算法的执行情况,请设置CrossoverFraction选项1.0然后重新运行求解器。
选项。CrossoverFraction = 1;[x, fval] = ga(有趣,据nvar ,[],[],[],[],[],[],[], 选项)

X = -0.0275 -0.0043 0.0372 -0.0118 -0.0377 -0.0444 -0.0258 -0.0520
优化终止:平均改变在的健身价值少比options.FunctionTolerance。X = 0.4014 0.0538 0.7824 0.1930 0.0513 -0.4801 0.9988 -0.0059 0.0875 0.0302 fval = 3.0843
在这种情况下,算法从初始种群中的个体中选择基因,并将它们重组。算法不能创造任何新的基因,因为没有突变。该算法在第8代生成它可以使用这些基因的最佳个体,在第8代,最佳适应度图成为水平。在此之后,它创造出最佳个体的新副本,然后将其选择为下一代。到第17代,种群中的所有个体都是相同的,即最佳个体。当这种情况发生时,个体之间的平均距离是0。由于算法在第8代之后不能提高最优适应度值,所以在50代之后停止,因为摊位代被设置为50.
突变没有交叉
要查看遗传算法在没有交叉时的执行情况,请设置CrossoverFraction选项0.
选项。CrossoverFraction = 0;[x, fval] = ga(有趣,据nvar ,[],[],[],[],[],[],[], 选项)

在这种情况下,算法应用的随机变化不会提高第一代最优个体的适应度值。虽然它改善了其他个体的个体基因,正如你在上面的图中通过适应度函数的平均值的下降所看到的,这些改进的基因永远不会与最佳个体的基因结合,因为没有交叉。因此,最佳适应度图为level,算法停留在第50代。
比较不同交叉分数的结果
这个例子deterministicstudy.m将遗传算法应用于拉斯特里金函数的结果与应用于拉斯特里金函数的结果进行比较CrossoverFraction选项设置为0,.2,.4,.6,.8,1.该示例运行了10代。在每一代中,该示例绘制出前几代的最佳适应度值的均值和标准差CrossoverFraction选择。
要运行示例,输入
deterministicstudy
在MATLAB®提示。示例完成后,绘图如下图所示。

下面的图显示了10代以上的最佳适应度值的平均值和标准差,每个值的交叉分数。上面的图用彩色编码显示了每一代的最佳适应度值。
对于这个适应度函数,设置交叉部分来0.8产生最好的结果。然而,对于另一个适应度函数,则有不同的设置交叉部分可能会产生最好的结果。

