PredictionConfidenceInterval
对象包含置信区间模型预测的结果
描述
的PredictionConfidenceInterval对象包含了置信区间模型预测的结果(即根据估计参数仿真结果)。
创建
创建一个置信区间预测对象使用sbiopredictionci。
属性
ResponseNames- - - - - -模型响应的名字
单元阵列的特征向量
这个属性是只读的。
模型反应参数适合的名字,指定为一个单元阵列的特征向量。每个单元格都包含响应的名字。
例子:{的中央。Drug_Central’} {' Peripheral.Drug_Peripheral '}
状态- - - - - -置信区间估计的状态
分类
这个属性是只读的。
置信区间估计状态,指定为以下分类值之一:
成功——适当的置信区间。也就是说,没有约束模型预测原始符合定义的参数范围。限制——置信区间被发现,但置信区间为模型反应是受到一个参数绑定中定义的原始。没有有价值的——不置信区间。可尊敬的——找到适当的置信区间,但其他的模型预测有一个估计的状态限制或没有有价值的。引导置信区间的状态总是设置为可尊敬的。
有关详细信息,请参见高斯置信区间计算模型预测和引导置信区间的计算。
例子:成功
类型- - - - - -置信区间类型
“高斯”|“引导”
这个属性是只读的。
置信区间类型,指定为“高斯”或“引导”。
例子:“引导”
α- - - - - -置信水平
积极的标量
这个属性是只读的。
信心水平,(1 -α)* 100%,指定为一个积极的标量在0和1之间。
例子:0.01
groupname- - - - - -从数据用于拟合原始组名称
单元阵列的特征向量
这个属性是只读的。
原始数据用于拟合模型的组名称,指定为一个单元阵列的特征向量。每个单元格都包含一组的名称。
例子:{' 1 '}{' 2 '}{' 3 '}
结果- - - - - -置信区间的结果
表
这个属性是只读的。
置信区间的结果,指定为一个表。表包含以下列。
| 列名 | 描述 |
|---|---|
| 集团 | 组名称 |
| 响应 | 模型响应的名字 |
| 时间 | 仿真时间 |
| 估计 | 估计响应值 |
| ConfidenceInterval | 置信区间值 |
ExitFlags- - - - - -在计算出口标志返回引导置信区间
向量
这个属性是只读的。
在计算出口标志返回引导置信区间,指定为一个整数向量。返回的每个整数都是退出标志(除了估计函数nlinfit在引导)用于合适的参数。相同的估计函数中使用原始适合用于引导。
每个国旗,表示拟合的成功或失败的状态执行创建一个引导样本。参考参考页面相应的估计函数退出标志的含义。
如果估计函数不返回退出国旗,ExitFlags被设置为[]。为高斯置信区间,ExitFlags不支持,总是设金宝app置为[]。
对象的功能
情节 |
情节置信区间模型预测的结果 |
例子
计算置信区间估计PK参数和模型预测
加载数据
加载示例数据符合。数据存储的表变量ID,时间,CentralConc,PeripheralConc。这种合成数据代表了等离子体浓度测量的时间进程在八个不同的时间点对中央和周边隔间后注入剂量3个人。
负载data10_32R.matgData = groupedData(数据);gData.Properties。VariableUnits = {”,“小时”,毫克/升的,毫克/升的};sbiotrellis (gData“ID”,“时间”,{“CentralConc”,“PeripheralConc”},“标记”,“+”,…“线型”,“没有”);

创建模型
创建一个两舱制模式。
pkmd = PKModelDesign;pkc1 = addCompartment (pkmd,“中央”);pkc1。DosingType =“注入”;pkc1。EliminationType =“linear-clearance”;pkc1。HasResponseVariable = true;pkc2 = addCompartment (pkmd,“外围”);模型=构造(pkmd);configset = getconfigset(模型);configset.CompileOptions。UnitConversion = true;
定义剂量
定义注入剂量。
剂量= sbiodose (“剂量”,“TargetName”,“Drug_Central”);剂量。开始时间= 0;剂量。数量= 100;剂量。率= 50;剂量。AmountUnits =毫克的;剂量。时间Units =“小时”;剂量。RateUnits =“毫克/小时”;
定义参数
定义的参数估计。设置参数为每个参数范围。除了这些明确的界限,参数转换(如日志、分对数或probit)对隐式边界。
responseMap = {“Drug_Central = CentralConc”,“Drug_Peripheral = PeripheralConc”};paramsToEstimate = {“日志(中央)”,的日志(外围),“12”,“Cl_Central”};estimatedParam = estimatedInfo (paramsToEstimate,…“InitialValue”(1 1 1),…“界限”,(0.1 - 3;0.1 10 0 10;0.1 - 2));
合适的模型
执行一个未共享健康,也就是说,一组为每个病人估计参数。
unpooledFit = sbiofit(模型、gData responseMap estimatedParam,剂量,“池”、假);
执行一个合并,即一组所有患者的估计参数。
pooledFit = sbiofit(模型、gData responseMap estimatedParam,剂量,“池”,真正的);
计算估计参数的置信区间
计算95%置信区间估计为每个参数未共享。
ciParamUnpooled = sbioparameterci (unpooledFit);
显示结果
显示表格格式的置信区间。对于细节每个估计状态的意思,明白了参数置信区间估计的状态。
ci2table (ciParamUnpooled)
ans = 12 x7表组名估计ConfidenceInterval类型α状态________ _____________ ________ _____ _____ * * *⒈1{“中央”}1.422 1.1533 1.6906 0.05高斯可尊敬的{‘外围’}1 1.5629 0.83143 2.3551 0.05高斯限制1{“12”}0.47159 0.20093 0.80247 0.05高斯限制1 {‘Cl_Central} 0.52898 0.44842 0.60955 0.05高斯可尊敬的2{“中央”}1.8322 1.7893 1.8751 0.05高斯成功2{‘外围’}5.3368 3.9133 6.7602 0.05高斯成功2{“12”}0.27641 0.2093 0.34351 0.05高斯成功2 {‘Cl_Central} 0.86034 0.80313 0.91755 0.05高斯成功3{“中央”}1.6657 1.5818 1.7497 0.05高斯成功3{‘外围’}5.5632 4.7557 6.3708 0.05高斯成功3{“12”}0.78361 0.65581 0.91142 0.05高斯成功3 {‘Cl_Central} 1.0233 0.96375 1.0828 0.05高斯成功
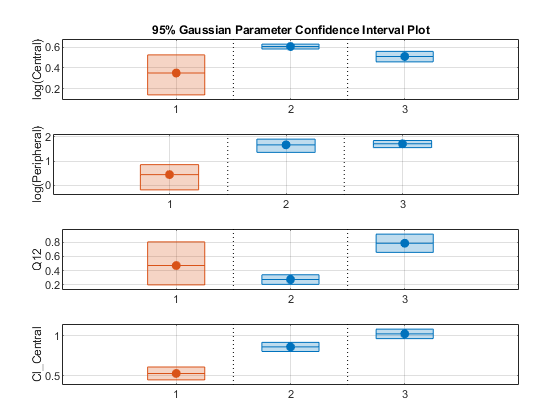
绘制置信区间。如果估计的置信区间成功蓝色,它是绘制(第一个默认颜色)。否则,它用红色(第二个默认颜色),这表明拟合参数可能需要进一步调查。如果置信区间没有有价值的,然后集中交叉功能地块红线。如果有任何改变参数估计值0(对数变换)和1或0 (probit或分对数变换),然后不绘制参数估计的置信区间。看到的颜色顺序,类型get(大的,“defaultAxesColorOrder”)。
组显示在相同的顺序从左到右,他们出现在groupname对象的属性,用于标签的轴。y标签转换后的参数名称。
情节(ciParamUnpooled)
计算池的置信区间。
ciParamPooled = sbioparameterci (pooledFit);
显示置信区间。
ci2table (ciParamPooled)
ans = 4 x7表组名估计ConfidenceInterval类型α状态________ _____________ ________ ________ * * * _____ ___________汇集{“中央”}1.6626 1.3287 1.9965 0.05高斯可尊敬的集合{‘外围’}2.687 0.89848 4.8323 0.05高斯约束集合{“12”}0.44956 0.11445 0.85152 0.05高斯约束集合{‘Cl_Central} 0.78493 0.59222 0.97764 0.05高斯可尊敬的
绘制置信区间。组名是贴上“池”,表示健康。
情节(ciParamPooled)

情节一起置信区间的结果。默认情况下,每个参数估计的置信区间是绘制在一个单独的轴。竖线组参数的置信区间估计,计算在公共健康。
ciAll = [ciParamUnpooled; ciParamPooled];情节(ciAll)

你也可以画上所有在一个轴组合参数估计的置信区间使用“分组”布局。
情节(ciAll“布局”,“分组”)

在这个布局,您可以指向每个置信区间的中心标记的组名。每个估计参数是由竖线分隔。垂直虚线组参数的置信区间估计,计算在一个共同的健康。参数范围中定义的原始适合方括号。注意不同尺度上的轴由于参数转换。例如,y轴的12个在线性范围内,但这的中央在对数尺度由于其对数变换。
计算模型预测的置信区间
为模型计算95%置信区间预测,也就是说,使用估计参数仿真结果。
%的汇集ciPredPooled = sbiopredictionci (pooledFit);%的未共享ciPredUnpooled = sbiopredictionci (unpooledFit);
图模型预测的置信区间
每组的置信区间是绘制在一个单独的列中,并且每个反应是绘制在一个单独的行。红色置信区间的限制范围。置信区间范围不限于用蓝色绘制。
情节(ciPredPooled)
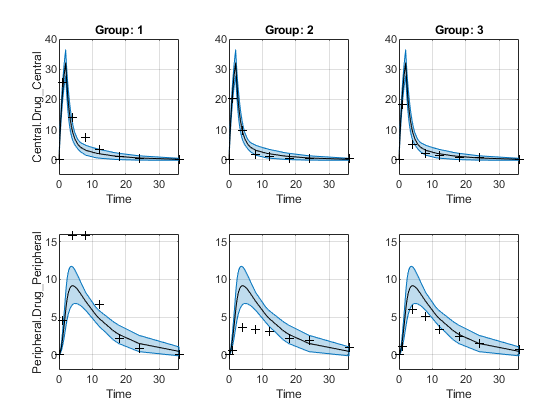
情节(ciPredUnpooled)

版本历史
介绍了R2017b
MATLAB命令
你点击一个链接对应MATLAB命令:
运行该命令通过输入MATLAB命令窗口。Web浏览器不支持MATLAB命令。金宝app

你也可以从下面的列表中选择一个网站:
表现最好的网站怎么走吗
选择中国网站(中文或英文)最佳站点的性能。其他MathWorks国家网站不优化的访问你的位置。