sbiopredictionci
计算模型预测的置信区间(需要统计和机器学习工具箱)
描述
ci= sbiopredictionci (fitResults)fitResults,一个NLINResults或OptimResults返回的sbiofit.ci是一个PredictionConfidenceInterval对象,该对象包含计算的置信区间数据。
ci= sbiopredictionci (fitResults,名称,值)名称,值对参数。
例子
计算估计PK参数和模型预测的置信区间
加载数据
加载样本数据以适应。数据存储为带有变量的表ID,时间,CentralConc,PeripheralConc.这一合成数据代表了三个人在输注剂量后,在中央和外周室的八个不同时间点测量的血浆浓度的时间过程。
负载data10_32R.matgData = groupedData(data);gData.Properties.VariableUnits = {”,“小时”,毫克/升的,毫克/升的};sbiotrellis (gData“ID”,“时间”,{“CentralConc”,“PeripheralConc”},“标记”,“+”,...“线型”,“没有”);

创建模型
创建一个两室模型。
pkmd = PKModelDesign;pkc1 = add隔间(pkmd,“中央”);pkc1。DosingType =“注入”;pkc1。EliminationType =“linear-clearance”;pkc1。HasResponseVariable = true;pkc2 = add隔间(pkmd,“外围”);Model = construct(pkmd);Configset = getconfigset(模型);configset.CompileOptions.UnitConversion = true;
定义剂量
确定输注剂量。
剂量= sbiodose(“剂量”,“TargetName”,“Drug_Central”);剂量。StartTime = 0;剂量。金额= 100;剂量。费率= 50;剂量。AmountUnits =毫克的;剂量。时间Units =“小时”;剂量。RateUnits =“毫克/小时”;
定义参数
定义要估计的参数。为每个参数设置参数边界。除了这些显式的边界之外,参数转换(如log、logit或probit)还施加了隐式的边界。
responseMap = {'Drug_Central = CentralConc','Drug_Peripheral = PeripheralConc'};paramsToEstimate = {“日志(中央)”,的日志(外围),“12”,“Cl_Central”};estimatedParam = estimatedInfo(paramsToEstimate,...“InitialValue”,[1 1 1 1 1],...“界限”,[0.1 3;0.1 10;0 10;0.1 2]);
合适的模型
执行未合并拟合,即对每个患者进行一组估计参数。
unpooledFit = sbiofit(模型,gData,responseMap,estimatedParam,剂量,“池”、假);
进行合并拟合,即对所有患者进行一组估计参数。
pooledFit = sbiofit(模型,gData,responseMap,estimatedParam,剂量,“池”,真正的);
计算估计参数的置信区间
计算未合并拟合中每个估计参数的95%置信区间。
ciParamUnpooled = sbioparameterci(unpooledFit);
显示结果
以表格格式显示置信区间。各估计状态的含义请参见参数置信区间估计状态.
ci2table (ciParamUnpooled)
ans = 12x7表组名估计置信区间类型Alpha状态_____ ______________ ________ __________________ _____________ ___________ 1 {'Central'} 1.422 1.1533 1.6906高斯0.05可估计1 {'Peripheral'} 1.5629 0.83143 2.3551高斯0.05约束1 {'Q12'} 0.47159 0.20093 0.80247高斯0.05约束1 {'Cl_Central'} 0.52898 0.44842 0.60955高斯0.05可估计2 {'Central'} 1.8322 1.7893 1.8751高斯0.05成功2 {'Peripheral'} 5.3368 3.9133 6.7602高斯0.05 success 2 {'Q12'} 0.27641 0.2093 0.34351高斯0.05 success 2 {'Cl_Central'} 0.86034 0.80313 0.91755高斯0.05 success 3 {'Central'} 1.6657 1.5818 1.7497高斯0.05 success 3 {'Peripheral'} 5.5632 4.7557 6.3708高斯0.05 success 3 {'Q12'} 0.78361 0.65581 0.91142高斯0.05 success 3 {'Cl_Central'} 1.0233 0.96375 1.0828高斯0.05 success
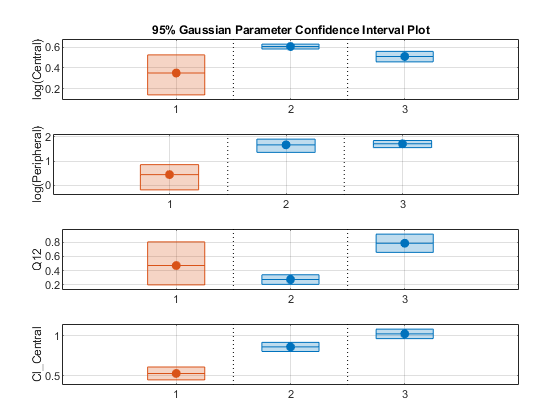
绘制置信区间。如果某个置信区间的估计状态为成功,它以蓝色(第一个默认颜色)绘制。否则,用红色(第二种默认颜色)表示,这表明可能需要对拟合参数进行进一步的研究。如果置信区间为没有有价值的,则该函数绘制一条带有居中十字的红线。如果有任何经过转换的参数,其估计值为0(对于log变换)和1或0(对于probit或logit变换),则不为这些参数估计值绘制置信区间。要查看颜色顺序,请键入get(大的,“defaultAxesColorOrder”).
组从左到右显示的顺序与它们在groupname对象的属性,该属性用于标记x轴。y标签是转换后的参数名。
情节(ciParamUnpooled)
计算合并拟合的置信区间。
ciParamPooled = sbioparameterci(pooledFit);
显示置信区间。
ci2table (ciParamPooled)
ans = 4x7表组名估计置信区间类型Alpha状态______ ______________ ________ __________________ _____________ ___________ pooled {'Central'} 1.6626 1.3287 1.9965高斯0.05估计的pooled {'Peripheral'} 2.687 0.89848 4.8323高斯0.05约束的pooled {'Q12'} 0.44956 0.11445 0.85152高斯0.05约束的pooled {'Cl_Central'} 0.78493 0.59222 0.97764高斯0.05估计
绘制置信区间。组名被标记为“pooled”以表示这种匹配。
情节(ciParamPooled)

把所有的置信区间结果画在一起。默认情况下,每个参数估计的置信区间在单独的轴上绘制。垂直线组参数估计的置信区间是在一个共同拟合中计算的。
ciAll = [ciParamUnpooled;ciParamPooled];情节(ciAll)

您还可以使用“分组”布局在参数估计分组的一个轴上绘制所有置信区间。
情节(ciAll“布局”,“分组”)

在此布局中,您可以指向每个置信区间的中心标记以查看组名。每个估计的参数由一条垂直的黑线分隔。垂直虚线组参数估计的置信区间是在一个共同拟合中计算的。在原始拟合中定义的参数边界用方括号标记。注意由于参数转换,y轴上的不同尺度。例如,的y轴12个是线性尺度,但中央是在对数尺度上,因为它是对数变换。
计算模型预测的置信区间
计算模型预测的95%置信区间,即使用估计参数的模拟结果。
%用于合并拟合ciPredPooled = sbiopredictionci(pooledFit);%用于未合并的拟合ciPredUnpooled = sbiopredictionci(unpooledFit);
绘制模型预测的置信区间
每个组的置信区间在单独的列中绘制,每个响应在单独的行中绘制。受边界限制的置信区间用红色表示。不受边界限制的置信区间用蓝色表示。
情节(ciPredPooled)
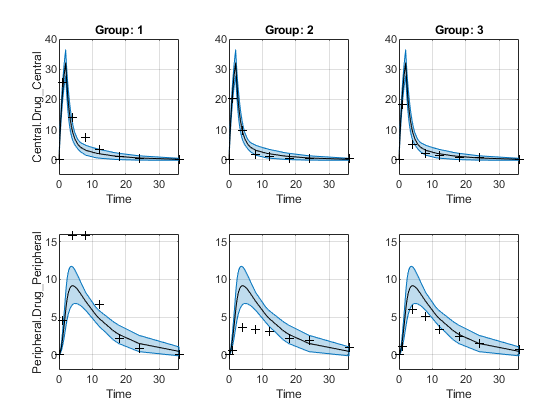
情节(ciPredUnpooled)

输入参数
fitResults- - - - - -参数估计结果sbiofit
NLINResults对象|OptimResults对象|向量
参数估计结果sbiofit,指定为NLINResults,OptimResults,或一个对象向量,用于从相同对象返回的未池拟合sbiofit调用。
名称-值参数
指定可选参数对为Name1 = Value1,…,以=家,在那里的名字参数名称和价值对应的值。名称-值参数必须出现在其他参数之后,但对的顺序无关紧要。
在R2021a之前,使用逗号分隔每个名称和值,并将其括起来的名字在报价。
例子:“阿尔法”,0.01,“类型”,“引导”指定使用引导方法计算99%置信区间。
α- - - - - -置信水平
0.05(默认)|积极的标量
信心水平,(1 -α) * 100%,指定为逗号分隔的对,由“α”在0到1之间是一个正标量。默认值为0.05,即计算95%置信区间。
例子:“阿尔法”,0.01
NumSamples- - - - - -用于自举的样本数量
1000(默认)|正整数
用于引导的样本数目,指定为逗号分隔的对,由“NumSamples”一个正整数。这个数字定义了在置信区间计算期间为生成自举样本而执行的拟合次数。数字越小,置信区间的计算速度就越快,但代价是精度降低。
例子:“NumSamples”,500年
显示- - - - - -返回到命令行的显示级别
“关闭”(默认)|“没有”|“最后一次”
返回到命令行的显示级别,由逗号分隔的对组成“显示”和一个字符向量。“关闭”(默认)或“没有”不显示输出。“最后一次”计算结束时显示一条消息。
例子:“显示”、“最后”
UseParallel- - - - - -并行计算置信区间的逻辑标志
真正的|假
并行计算置信区间的逻辑标志,指定为逗号分隔的对,由“UseParallel”而且真正的或假.默认情况下,使用原始拟合中的并行选项。如果此参数设置为真正的和并行计算工具箱™可用时,将忽略原始拟合中的并行选项,并并行计算置信区间。
对于高斯置信区间:
如果输入
fitResults是结果对象的向量,则对每个对象的置信区间进行并行计算。高斯置信区间计算起来很快。因此,并行化原始拟合可能更有益(sbiofit)和未设置UseParallel为真sbiopredictionci.
对于Bootstrap置信区间:
该函数将
UseParallel旗帜bootci.结果对象的输入向量上没有并行化。
请注意
如果你有一个用于随机数生成的全局流,它有许多子流,可以以可重复的方式并行计算,sbiopredictionci首先检查工人的数量是否与子流的数量相同。如果是,则函数集UseSubstreams来真正的在statset选项并传递给bootci(统计和机器学习工具箱).否则,默认情况下将忽略子流。
例子:“UseParallel”,真的
输出参数
ci-置信区间结果
PredictionConfidenceInterval对象
置信区间结果,返回为aPredictionConfidenceInterval对象。对于未合并的不适,ci可以是向量PredictionConfidenceInterval对象。
更多关于
模型预测的高斯置信区间计算
模型围绕参数估计线性化P美国东部时间由返回的拟合结果获得的sbiofit.的CovarianceMatrix使用线性化模型进行转换。此外,隐式参数边界(日志,probit,或分对数在原始拟合中指定的参数变换)和显式参数边界(如果在原始拟合中指定)也通过线性化模型进行映射。
为了使模型线性化,sbiopredictionci首先检查灵敏度分析功能是否在原始配合中打开。如果特征是打开的,则函数使用通过复阶跃微分计算的雅可比矩阵。如果特征是关闭的,雅可比矩阵计算使用有限差分。有限差分可能是不准确的,并考虑打开敏感性分析运行时的特性sbiofit.
函数使用转换后的CovarianceMatrix并计算每个时间步估计模型响应的高斯置信区间。
在置信区间受到原始拟合中定义的参数边界约束的情况下,根据Wu, H.和Neale, M.描述的方法调整置信区间边界。[1].
对于每个模型响应,函数首先确定置信区间是否无界。如果是,则将相应模型响应的估计状态设置为
没有有价值的.否则,如果响应的置信区间受到原始拟合中定义的参数界限的约束,则函数将其状态设置为
限制.参数转换(例如日志,probit,或分对数)对估计的参数施加隐式边界,例如正性约束。这样的边界可能导致置信度的高估,即置信区间可能小于预期。如果没有置信区间有估计状态
没有有价值的或限制,则函数将所有模型响应的估计状态设置为成功.否则,其余模型响应的估计状态设为可尊敬的.
自举置信区间计算
的bootci(统计和机器学习工具箱)函数从统计和机器学习工具箱™用于计算bootstrap置信区间。第一个输入nboot为样本数(NumSamples),以及第二个输入bootfun是执行这些操作的函数。
重新采样数据(如果有多个组,则在每个组中独立进行)。
运行一个符合重采样数据的参数。
利用估计的参数对模型进行仿真,得到模型响应。
返回模型响应。
估计状态总是设置为可尊敬的由于函数不能确定置信区间是否受到参数估计的边界的约束。
参考文献
[1]吴,H.和M.C.尼尔。有界参数的调整置信区间行为遗传学.42 (6), 2012, pp. 886-898。
扩展功能
自动平行支撑金宝app
通过使用并行计算工具箱™自动并行运行计算来加速代码。
平行运行,设置“UseParallel”来真正的.
有关更多信息,请参见“UseParallel”名称-值对参数。
版本历史
在R2017b中引入
MATLAB命令
你点击了一个对应于这个MATLAB命令的链接:
在MATLAB命令窗口中输入该命令来运行该命令。Web浏览器不支持MATLAB命令。金宝app

您也可以从以下列表中选择一个网站:
如何获得最佳的网站性能
选择中国站点(中文或英文)以获得最佳站点性能。其他MathWorks国家站点没有针对您所在位置的访问进行优化。