PK / PD建模和仿真指导抗生素给药策略
该实施例显示了如何对抗菌剂进行药代动力学/药物动力学(PK / PD)模型的蒙特卡罗模拟。这个例子是从Katsube等人的调整。[1]此示例还显示了如何使用Simbiology®simfunction.对象并行执行参数扫描。
此示例需要统计和机器学习工具箱™。如果您有并行计算工具箱™软件,则可以提高性能。
背景
Katube等人。[1]使用PK / PD建模和仿真方法来确定多翅膜,碳烯烯抗生素的最有效的剂量方案。他们的研究目标是:
开发PK / PD模型,描述Doripenem对几种假单胞菌铜绿假单胞菌菌株的抗菌作用
使用Monte Carlo模拟来比较四种常见的抗生素剂量制度的疗效,并确定最有效的给药策略
探讨肾功能对治疗抗菌疗效的影响
在该示例中,我们将实施由Katsube等人开发的抗菌PK / PD模型。[1]在SimBiology®中,并复制他们工作中描述的蒙特卡罗模拟的结果。
参考
[1] T. Katsube,Y. Yano,T. Wajima,Y. Yamano和M. Takano。药代动力学/药效学建模和模拟确定多翅膜的有效剂量方案。医药科学(2010)99(5),2483-91。
PK / PD模型
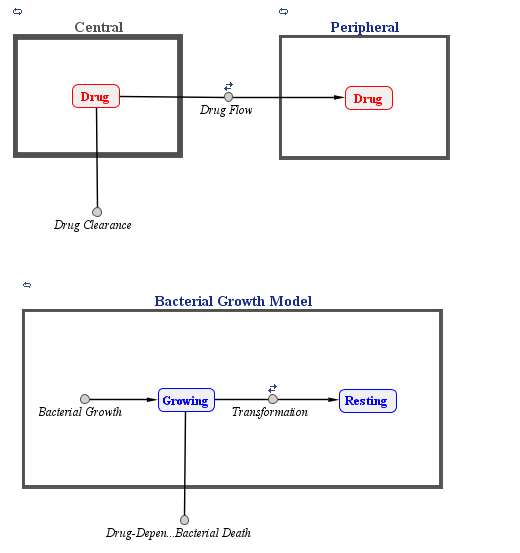
Katube等人。假设具有从中央隔间的线性消除的双隔室输液模型,以描述多翅膜的药代动力学。对于细菌生长模型,他们认为总细菌群由药物易感生长细胞和药物不敏感静息细胞组成。通过简单的Emax型模型将药物的抗菌作用包含在细菌的杀伤率中:
![$$杀戮率= \ frac {kmax * [drug] * [growing]} {kc50 + [drug]} $$](http://www.tatmou.com/help/examples/simbio/win64/antibioticdemo_eq06411219986050949670.png)
在哪里[药品]是中央隔室中药物的浓度(UG / ml),[生长]是CFU / ml(CFU =菌落形成单位)中生长细菌群的计数。kmax.是最大杀伤率常数(1 /小时)和KC50.是michaelis-menten率常数(Ug / ml)。
模型的偶像学实现的图形视图如下所示。
%加载模型sbioloadproject('antiaLatialpkpd.sbproj'那'm1');
剂量方案
Katube等人。使用四种常见的抗生素剂量策略模拟模型。
每天250毫克两次(B.I.D.)
每天250毫克三次(T.I.D.)
每天两次(B.I.D.)500毫克
每天三次(T.I.D.)500毫克
输注剂量在所有四种剂量方案中使用,输注时间设定为30分钟。在血症学中,这些剂量方案已被实施为剂量物体。
%选择模型中的剂量对象Dosenames = {'250 mg'那'250 mg tid'那'500 mg bid'那'500 mg tid'};为了IdoseGrp = 1:长度(Dosenames)doseregimens(Idosegrp)= SbioSelect(M1,'姓名',Dosenames {idosegrp});结尾
虚拟人群的描述
基于人口统计变量和PK / PD参数的分布生成了个体的虚拟群体。分布的类型和分布参数的值基于来自日本在日本的Doripenem的早期临床试验的数据。
注意:在[1]中,在每个剂量组中模拟5,000名虚拟患者。在这个例子中,我们将在每组中使用1,000名患者。为了模拟不同的人口大小,改变价值纳育以下。
% 设置npatients = 1000;每剂量组%的患者数量ndosegrps = 4;%有效的剂量方案数量
人口统计变量的分布:
重量 (WT.)和年龄(年龄)分别从正常分布中取样,平均分别为51.6千克和71.8岁,分别为11.8千克和11.9岁。26%的人口被认为是女性。血清肌酐水平(克拉)从逻辑正式分布中取样,典型值(几何平均值)为0.78mg / dl,变异系数(cv)为32.8%。肌酐清除率(CRCL.)使用Cockcroft-Gault方程计算。
输入的输入lognrnd.功能是平均值(亩)标准偏差(Sigma.)相关的正态分布。在这里和整个例子中,亩和Sigma.根据报告的典型值和伐诺型分布的变异系数计算。您可以使用以下定义来计算它们。看看lognstat.有关更多信息的文档。
mu = @(m,v)日志(m ^ 2 / sqrt(v + m ^ 2));sigma = @(m,v)sqrt(log(v / m ^ 2 + 1));m = @(typicalValue)typicalValue;v = @(typicalValue,cv)typicalvalue ^ 2 * cv ^ 2;%患者人口统计学RNG('默认');wt = normrnd(51.6,11.8,幼儿,ndosegrps);%单位:kg年龄= Normrnd(71.8,11.9,幼儿,Ndosegrps);%单位:年份scr_mu = mu(m(0.78),v(0.78,0.328));scr_sigma = sigma(m(0.78),v(0.78,0.328));scr = lognrnd(scr_mu,scr_sigma,npatients,ndosegrps);%单位:ml /分钟%性别比例ID = 1:npatiants * ndosegrps;IDFemale = RandSample(ID,ROUND(0.26 * NdoseGRPS * npatiants));%26%的女性
肌酐清除(使用Cockcroft-Gault方程)
crcl =(140岁)。* wt./(scr*72);%单位:ml /分钟CRCL(IDFemale)= CRCL(IDFemale)* 0.85;%乘以女性0.85
药代动力学(PK)参数的分布:
PK参数,中央那K12., 和K21,从逻辑正式分布中取样,分别具有7.64升的典型值,1.59 1 /小时和2.26 1 /小时,以及20%的变异系数(CV)。中央是中央隔间的分配量,以及K12.和K21是转移率常数之间的中央和外围隔间。
central_mu = mu(m(7.64),v(7.64,0.20));Central_Sigma = Sigma(M(7.64),v(7.64,0.20));K12_mu = mu(m(1.59),v(1.59,0.20));K12_sigma = Sigma(M(1.59),v(1.59,0.20));K21_mu = mu(m(2.26),v(2.26,0.2));K21_sigma = Sigma(M(2.26),V(2.26,0.2));Central = lognrnd(Central_Mu,Central_Sigma,Npatiants,Ndosegrps);%单位:升K12 = lognrnd(k12_mu,k12_sigma,npatients,ndosegrps);%单位:1 /小时k21 = lognrnd(k21_mu,k21_sigma,npatients,ndosegrps);%单位:1 /小时
药物清除率,CL.,假设通过以下等式依赖于群体清除率的线性:
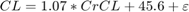
在哪里 是从正态分布中采样的添加剂残留误差,平均值为0 ml /分钟和22ml /分钟的标准偏差。
是从正态分布中采样的添加剂残留误差,平均值为0 ml /分钟和22ml /分钟的标准偏差。
CL = 1.07 * CRCL + 45.6 + NOMARND(0,22,幼儿,NDOSEGRS);%单位:ml /分钟
药效学(PD)参数的分布:
生长休息的转化率常数,K1和K2,用典型值为5.59E-5和0.02971 /小时的典型值取样,每次具有20%的CV。kmax.从逻辑正式分布中取样,典型值为3.5 1 /小时和15.9%CV。
K1_mu = Mu(m(5.59e-5),v(5.59e-5,0.2));K1_SIGMA = SIGMA(M(5.59e-5),V(5.59e-5,0.2));K2_mu = mu(m(0.0297),v(0.0297,0.2));K2_sigma = Sigma(m(0.0297),v(0.0297,0.2));kmax_mu = mu(m(3.50),v(3.50,0.159));kmax_sigma = sigma(m(3.50),v(3.50,0.159));k1 = lognrnd(k1_mu,k1_sigma,npatiants,ndosegrps);%单位:1 /小时k2 = lognrnd(k2_mu,k2_sigma,npatients,ndosegrps);%单位:1 /小时kmax = lognrnd(kmax_mu,kmax_sigma,npatients,ndosegrps);%单位:1 /小时
Katube等人。假设价值观K1那K2和kmax.与被治疗的细菌菌株无关。的价值bet,净生长速率恒定,固定在1.5 1 /小时。
基于几种菌株的实验,作者得出结论是价值KC50.通过以下等式线性地依赖于细菌菌株的最小抑制浓度(MIC)。
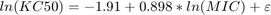
在哪里 是从正态分布采样的添加剂残留误差,其平均值为0和标准偏差为1.06 ug / ml。在模拟中,
是从正态分布采样的添加剂残留误差,其平均值为0和标准偏差为1.06 ug / ml。在模拟中,麦克风从离散分布中取样值,以及KC50.计算所选值的值麦克风使用上述等式。
基于71p.铜绿假单胞菌菌株的MIC值的百分比分布MicValue = [0.0625,0.125,0.25,0.5,1,2,4,8,16,32];MICFREQ = [5,8,9,14,7,8,9,5,2,4];使用RANDSAMPLE的离散分布的%样本MIC值麦克风=南(Npatiants,Ndosegrps);%preallocate为了Idosegrp = 1:ndosegrps mic(:, idosegrp)= randsample(micvalue,npatients,true,micfreq);结尾KC50 = EXP(-1.91 + 0.898 * log(麦克风)+ 1.06 * RANDN(纳氏,NDOSEGRPS));%单位:微克/毫升
仿真设置与设计
创建一个simfunction.允许您在并行执行模型模拟和参数扫描的对象。在此示例中,您将改变8个参数,中央那K12.那K21那CL.那K1那K2那kmax., 和KC50.。选择生长和休息的细菌数量,生长和休息作为响应,即您想要观察的仿真结果,同时改变这些输入参数。
选择要不同的参数。
params = {'中央'那'K12'那'k21'那'Cl'那'k1'那'k2'那'kmax'那'kc50'};
选择响应。
观察到= {'[细菌生长模型] .gring'那......'[细菌生长模型]。resting'};
设置模板剂量。
tempdose = sbiodose('剂量');tempdose.target =.'central.drug';tempdose.amountunits ='毫克';tempdose.timeUnits ='小时';tempdose.dentionParameTername =.'tdose';
创建一个simfunction.目的。放使用指α.为the启用并行计算。
simfunc = createImfunction(m1,params,观察到,tempdose,'使用指平行',真的);
创建输入矩阵披对于每种剂量组。
phi = cell(1,ndosegrps);为了i = 1:ndosegrps phi {i} = [central(:,i),k12(:,i),k21(:,i),......CL(:,i),k1(:,i),k2(:,i),......kmax(:,i),kc50(:,i)];结尾
集群计算
此示例使用预先配置到本地计算机的本地群集配置文件。您还可以搜索在AmazonEC2®上运行的其他MATLAB®ParrateServer™群集。在这一点家选项卡在环境部分,选择平行线>发现群集。要访问这些群集,您必须提供MathWorks®帐户登录信息。有关详细信息,请参阅发现群集并使用群集配置文件(并行计算工具箱)。
如果没有存在,则创建一个并行池。
如果缺乏(GCP)Parpool;结尾
使用“本地”配置文件启动并行池(Parpool)连接到并行池(工人数:6)。
对于所有剂量方案,模拟模型直到第一剂量的T = 2周。总细菌计数CFU每24小时(每天一次)对剂量方案的整个持续时间进行排序。
TOB = 0:24:336;% 小时ntpoints = length(tobs);%的抽样点数
严重感染患者的蒙特卡罗模拟
可以使用不同的PK / Pd索引测量药物的抗菌效果。Katube等人。设置细菌消除的标准log10(cfu)<0,CFU是总细菌计数。测量每种剂量方案的功效作为在剂量组中实现成功标准的群体的级分。这个功效度量,Pr {log10(cfu)<0},被跟踪为每种剂量组的时间函数。
在他们的模拟研究中,作者研究了剂量方案对两类患者的疗效:
中等感染(初始细菌计数= 1E4 CFU / mL)
严重感染(初始细菌计数= 1E7 CFU / mL)
在此示例中,我们将仅对严重的感染案件复制结果。注意,通过改变细菌数量的初始量(该方法,您可以轻松模拟其他情况,中度感染的患者(生长物种),在模型中至1E4 CFU / mL。
%preallocatecfu = nan(Ntpoints,npatiants);log10cfu = cell(1,ndosegrps);为了i = 1:ndosegrps disp(['模拟小组',num2str(i),'...'])%直接从每个现有剂量对象获取剂量表%给药方案。剂量= gettable(doseregimens(i));%模拟simdata = simfunc(phi {i},[],装饰,tobs);每个患者的生长和休息细菌计数的百分比为了j = 1:幼崽cfu(:,j)= sum(simdata(j).data,2);结尾%存储每个剂量组的数值数转换计数。log10cfu {i} = log10(cfu);结尾%保存结果log10cfu_250bid = log10cfu {1};log10cfu_250tid = log10cfu {2};log10cfu_500bid = log10cfu {3};log10cfu_500tid = log10cfu {4};
模拟组1 ...仿真组2 ...模拟组3 ...模拟组4 ...
关闭并行池。
删除(GCP('noicreate'));
细菌计数的时间课程
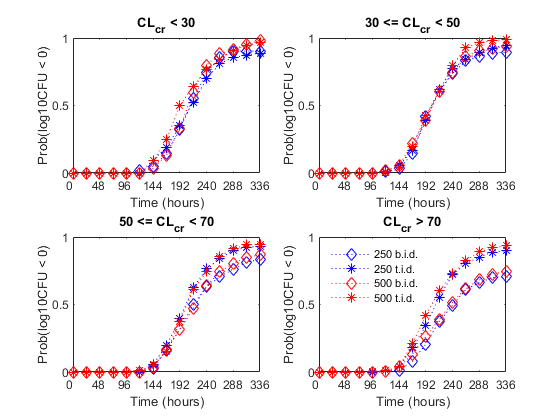
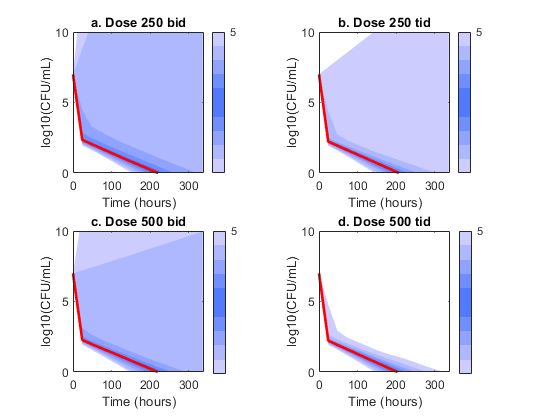
我们绘制了中位数(以红色)和百分位数(阴影)轮廓log10(cfu)所有四种剂量方案的水平。观察到,在所有四组中,中值时间课程型材表明,在治疗期结束前(336小时)之前,细菌根除是完整的。然而,从较高百分位的轮廓明显看出,治疗对于所有患者不成功。第95和第90百分位分布还表明,用较高频率(250坦)的较低量的较低量比具有更高量(500bid)的频率更低的剂量更有效。
hax1(1)=子图(2,2,1)plotcfucount(tobs,log10cfu_250bid,'一种。剂量250出价')hax1(2)=子图(2,2,2)plotcfucoun(tobs,log10cfu_250tid,'b。剂量250 tid')HAX1(3)=子图(2,2,3)PLOTCFUCOUNT(TOB,LOG10CFU_500BID,'C。剂量500出价')HAX1(4)=子图(2,2,4)PLOTCFUCOUNT(TOB,LOG10CFU_500TID,'d。剂量500 TID')%链接子图轴Linkaxes(HAX1)
hax1 =具有属性的轴:xlim:[0 1] ylim:[0 1] Xscale:'Linear'yScale:'Linear'GridlineStyle:' - '位置:[0.1300 0.5838 0.3347 0.3412]单位:'标准化'使用开始显示所有属性hax1 = 1×2轴阵列:轴轴hax1 = 1×3轴阵列:轴轴轴hax1 = 1×4轴阵列:轴轴轴轴
肾功能对抗菌活性的影响
最后,作者将剂量方案的功效谱与肾功能的函数进行了比较。他们根据肌酐清除率将患者分为四个肾功能群体(CRCL.):
肌酐清除群1:
CRCL.<30肌酐清除率组2:30 <=
CRCL.<50肌酐清关组3:50 <=
CRCL.<70肌酐清除群4:
CRCL.> = 70.
下图显示了肾功能(肌酐清除率)对四种剂量方案的抗菌效果的影响。观察到正常肾功能组(CRCL.> = 70),四种治疗策略的功效谱彼此显着不同。在这种情况下,500 mg t.i.d.剂量比其他方案更有效。相比之下,涉及肾功能障碍患者的模拟(CRCL.<30和30 <=CRCL.<50),我们没有看到治疗组之间的差异很大。这表明对于肾功能不全的患者,较小或更少的频率给药策略几乎可以使用,以及具有更高频率或给药量的计量策略。
%preallocateidcrclgrp = false(npatients,ndosegrps);%线条风格ls = {'BD:'那'B *:'那'rd:'那'r *:'};tittestr = {'cl_c_r <30'那......'30 <= cl_c_r <50'那......'50 <= cl_c_r <70'那......'cl_c_r> 70'};f =图;F.Color =.'W'为了ICRCLGRP = 1:4%肌酐清除群HAX2(ICRCLGRP)=子图(2,2,ICRCLGRP);标题(titlestr {icrclgrp});ylabel('prob(log10cfu <0)');Xlabel('时间(小时)');结尾%设置轴属性套(HAX2,'xtick',0:48:336,......'xticklabel',0:48:336,......'ylim',[0 1],......'xlim',[0 336],......'nextplot'那'添加'那......'盒子'那'在');肾功能组的%绘图结果:为了IdoseGrp = 1:ndosegrps%提取肾功能的指标idcrclgrp(:,1)= CRCL(:,IDosegrp)<30;IDCRCLGRP(:,2)= CRCL(:,IDosegrp)> = 30&CRCL(:,IDosegrp)<50;IDCRCLGRP(:,3)= CRCL(:,IDosegrp)> = 50&CRCL(:,IDosegrp)<70;IDCRCLGRP(:,4)= CRCL(:,IDosegrp)> = 70;为了ICRCLGRP = 1:4%肌酐清除群%计算概率pr = sum((log10cfu {idosegrp}(:, idcrclgrp(:, icrclgp)')<0),2)/ sum(idcrclgrp(:,iCrclgrp));% 阴谋绘图(HAX2(ICRCLGRP),TOB,PR,LS {IDOSEGRP},'Markersize'7)结尾结尾传奇(HAX2(4),{'250 b.i.d'那'250 t.i.d.'那'500 b.i.d.'那'500 t.i.d.'} ) 传奇地点西北传奇Boxoff.Linkaxes(HAX2)
f =图(1)具有属性:Number:1名称:''颜色:[1 1 1]位置:[680 678 560 420]单位:'像素'使用可以显示所有属性