模拟葡萄糖-胰岛素反应
本示例展示了如何在SimBiology®中使用基于正常和糖尿病人葡萄糖-胰岛素系统的生理学模型来模拟和分析模型。
这个例子需要统计和机器学习工具箱™和优化工具箱™。
参考文献
葡萄糖-胰岛素系统的膳食模拟模型。c·达拉·曼,r·a·里扎,c·科贝利。IEEE生物医学工程汇刊(2007) 54(10), 1740-1749。
口服葡萄糖吸收系统模型:金标准数据验证。达拉曼,卡米列里,科贝利。IEEE生物医学工程汇刊(2006) 53(12), 2472-2478。
目标
实现葡萄糖-胰岛素反应的SimBiology模型。
模拟正常和受损(糖尿病)受试者对一餐或多餐的葡萄糖-胰岛素反应。
使用以下方法执行参数估计
sbiofit用强制函数策略。
背景
在他们2007年的出版物中,Dalla Man等人开发了一个餐后人体葡萄糖-胰岛素反应的模型。该模型用常微分方程描述了系统的动力学。作者使用他们的模型来模拟一顿或多顿饭后的葡萄糖-胰岛素反应,包括正常的人类受试者和各种胰岛素损伤的人类受试者。损伤用交替的参数值和初始条件表示。
我们实现了SimBiology模型,m1由:
将Dalla Man et al.(2007)中的模型方程转化为反应、规则和事件。
将模型组织成两个隔间,一个是葡萄糖相关的物种和反应(命名为
葡萄糖的外表)和一个关于胰岛素相关的物种和反应(命名为胰岛素分泌).利用模型方程及表1和图1中的参数值和初始条件。
包括在Dalla Man等人(2006)中提出的胃排空率方程。
根据Dalla Man等人(2007)的规定,设置所有物种、隔间和参数的单位,从而允许使用单位转换来模拟SimBiology模型。(注意SimBiology也支持使用无量纲参数,通过设金宝app置它们的
ValueUnits财产无量纲的)。设置配置集
TimeUnits来小时,因为模拟是在7或24小时内进行的。以1公斤体重为基础,对原模型中以体重归一化的物种和参数进行变换。按照SimBiology的要求,这样做就形成了物种数量或浓度单位。
我们将SimBiology模型中的胰岛素损伤表示为具有以下名称的变体对象:
2型糖尿病
胰岛素敏感性低
高β细胞反应
β细胞反应性低
高胰岛素敏感性
我们将SimBiology模型中的膳食表示为剂量对象:
一种名为
一顿简单的饭菜表示模拟开始时一餐78克葡萄糖。一种名为
日常生活代表一天的膳食,相对于从午夜开始的模拟:早餐在8小时的模拟时间(上午8点)中含有45克葡萄糖,午餐在12小时(中午)中含有70克葡萄糖,晚餐在20小时(晚上8点)中含有70克葡萄糖。
SimBiology模型示意图如下所示:

设置
加载模型。
sbioloadproject (“insulindemo”,“m1”)
抑制在模拟过程中发出的信息警告。
warnSettings =警告(“关闭”,“SimBiology: DimAnalysisNotDone_MatlabFcn_Dimensionless”);
模拟正常人的葡萄糖-胰岛素反应
选择一顿简单的饭菜剂量对象并显示其属性。
mealDose = sbioselect(m1,“名字”,“一餐”);get (mealDose)
ans =带字段的结构:数量:78间隔:0速率:0 RepeatCount: 0 StartTime: 0活动:0 AmountUnits: 'gram' DurationParameterName: " EventMode: 'stop' LagParameterName: " RateUnits: " TargetName: 'Dose' TimeUnits: 'hour' Name: 'Single Meal' Parent: [1x1 SimBiology.]注释:" Tag: " Type: 'repeatdose' UserData: []
模拟7小时。
Configset = getconfigset(m1,“活跃”);configset。StopTime=7;
显示模拟时间单位(和StopTime单位)。
configset。TimeUnits
Ans =“小时”
模拟一个正常人的一顿饭。
normalMealSim = sbiosimulation (m1, configset, [], mealDose);
模拟2型糖尿病患者的葡萄糖-胰岛素反应
选择2型糖尿病变异并显示其特性。
糖尿病var = sbioselect(m1,“名字”,“2型糖尿病”)
diabticvar = SimBiology变体- 2型糖尿病(不活跃)内容索引:类型:名称:属性:值:1参数血浆容量…取值1.49 2参数k1取值0.042 3参数k2取值0.071 4参数血浆体积…参数值0.04 5参数m1参数值0.379 6参数m2参数值0.673 7参数m4参数值0.269 8参数m5参数值0.0526 9参数m6参数值0.8118 10参数取值0.6 11参数kmax取值0.0465 12参数kmin取值0.0076 13参数kbs取值0.023 14参数kgri取值0.0465 15参数f取值0.9 16参数a取值6e-05 17参数b取值0.68 18参数c取值0.00023 19参数d取值0.09 20参数胃Glu Af…取值125 21参数kp1取值3.09 22参数kp2取值0.0007 23参数kp3取值0.005 24参数kp4取值0.0786 25参数ki取值0.0066 26参数[Ins Ind Glu U…]取值1 27参数Vm0值4.65 28参数Vmx值0.034 29参数Km值466.21 30参数p2U值0.084 31参数K值0.99 32参数alpha值0.013 33参数beta值0.05 34参数gamma值0.5 35参数ke1值0.0007 36参数ke2值269 37参数基础等离子体G…值164.18 38参数基底血浆I…价值54.81
模拟2型糖尿病患者的一顿饭。
diabticmealsim = sbiosimulation (m1, configset, diabeticVar, mealDose);
比较模拟中最重要输出的结果。
血糖(种类)
血浆Glu Conc)血浆胰岛素(种类)
等离子体)内源性葡萄糖产生(参数
Glu刺激)出现率(参数
谷氨酸出现率)葡萄糖利用率(参数
Glu跑龙套)胰岛素分泌(参数
Ins Secr)
outputNames = {“Plasma Glu Conc”,《等离子体》,“Glu刺激”,...“Glu出现率”,“Glu Util”,“Ins Secr”};图;为i = 1: number (outputNames) subplot(2,3, i);[tNormal, yNormal] = normalMealSim.selectbyname(outputNames{i});[tDiabetic, yDiabetic] = diabticmealsim .selectbyname(outputNames{i});plot(tNormal, yNormal,“- - -”,...糖尿病患者,糖尿病患者,“——”);标注数字outputParam = sbioselect(m1,“名字”我,outputNames {});标题(outputNames{我});包含(的时间(小时));如果比较字符串(outputParam。类型,“参数”) ylabel (outputParam.ValueUnits);其他的ylabel (outputParam.InitialAmountUnits);结束xlim ([0 7]);%添加图例如果I == 3 legend({“正常”,“糖尿病”},“位置”,“最佳”);结束结束
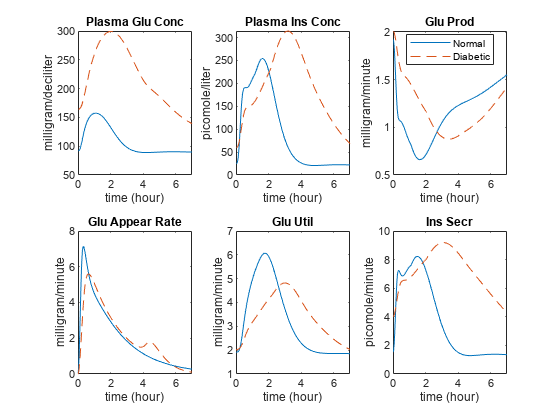
注意血浆中葡萄糖和胰岛素的浓度高得多,以及葡萄糖利用和胰岛素分泌的持续时间延长。
模拟一个正常人一日三餐
设置模拟StopTime到24小时。
configset。StopTime=24;
选择每日膳食剂量。
dayDose = sbioselect(m1,“名字”,“日常生活”);
模拟一个正常人的三顿饭。
normalDaySim = sbiosimulation (m1, configset, [], dayDose);
模拟残疾受试者一日三餐
模拟下列损伤组合:
损伤1:胰岛素敏感性低
损伤2:β细胞反应性高的损伤1
损伤3:β细胞反应性低
损伤4:胰岛素敏感性高的损伤3
将损伤存储在单元格数组中。
受损vars {1} = sbioselect(m1,“名字”,“胰岛素敏感性低”);受损vars{2} =[受损vars {1},]...sbioselect (m1,“名字”,“高β细胞反应”));受损vars {3} = sbioselect(m1,“名字”,“β细胞反应性低”);受损vars{4} =[受损vars {3},...sbioselect (m1,“名字”,“胰岛素敏感性高”));
模拟每种损伤。
为i = 1:4 impairment sims (i) = sbiosimulate(m1, configset, impairment vars {i}, dayDose);结束
比较血浆葡萄糖和胰岛素结果。
图;outputNames = {“Plasma Glu Conc”,《等离子体》};legendLabels = {{“正常”},...{“ins =β\ ',“ins +β\”},...{“β= Ins - \”,“+ Ins -β\”}};yLimits = [80 240;0 500];为i = 1:num (outputNames) [tNormal, yNormal] = selectbyname(normalDaySim, outputNames{i});[tImpair, y损害]= selectbyname(损伤sims, outputNames{i});图正常百分比Subplot (2,3,3 *i-2);情节(tNormal yNormal,“b -”);xlim (24 [0]);ylim (yLimits(我,:));包含(的时间(小时));传奇(legendLabels {1},“位置”,“西北”);% Plot低胰岛素Subplot (2,3,3 *i-1);情节(tImpair {1}, yImpair {1},“g——”, tImpair{2}, ycripple {2},“:”);xlim (24 [0]);ylim (yLimits(我,:));包含(的时间(小时));传奇(legendLabels {2},“位置”,“西北”);标题(outputNames{我});%图低BetaSubplot (2,3,3 *i);情节(tImpair {3}, yImpair {3},c -。, tImpair{4}, ycripple {4},“m -”);xlim (24 [0]);ylim (yLimits(我,:));包含(的时间(小时));传奇(legendLabels {3},“位置”,“西北”);结束
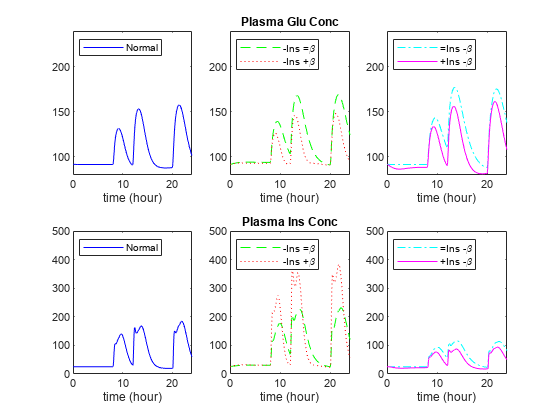
注意,低胰岛素敏感性(绿色虚线, )或低β细胞敏感性(虚线青色线, )导致血糖浓度升高和延长(图上排)。一种系统的低灵敏度可由另一种系统的高灵敏度部分补偿。例如,低胰岛素敏感性和高β细胞敏感性(虚线红色, )结果血糖浓度相对正常(图上排)。然而,在这种情况下,产生的血浆胰岛素浓度极高(图的底部)。
参数估计方法
作者没有同时估计整个模型的参数,而是使用强制函数策略对模型的不同子系统进行参数估计。这种方法需要额外的实验数据作为子模型的“输入”。在拟合过程中,输入数据决定了输入物种的动态。(在完整的模型中,输入的动态由微分方程确定。)在SimBiology术语中,您可以将强制函数实现为重复分配规则,该规则控制作为模型子系统输入的物种或参数的值。在下面的部分中,我们将使用SimBiology的参数拟合功能来细化作者报告的参数值。
葡萄糖外观胃肠道模型的拟合nlinfit
胃肠道模型代表了一顿饭中的葡萄糖是如何通过胃、肠道和肠道运输,然后被吸收到血浆中。这个子系统的输入是一顿饭中葡萄糖的含量,输出是血浆中葡萄糖的出现率。然而,由于作者报告的数值与参数和模拟结果不一致,我们也估计了膳食尺寸。因为这个输入只在模拟开始时出现,所以不需要强制函数。
这个函数sbiofit金宝app支持SimBiology模型中的参数估计,使用MATLAB™,统计和机器学习工具箱,优化工具箱和全局优化工具箱中的几种不同算法。首先,使用统计和机器学习工具箱功能估计参数nlinfit.
加载实验数据fitData = groupedData(可读数据(“GlucoseData.csv”,“分隔符”,”、“));在数据上设置单位fitData.Properties.VariableUnits = {...“小时”,...%时间单位毫克/分钟的,...%谷氨酸单位毫克/分升的,...% PlasmaGluConc单位毫克/分钟的,...% GluUtil单位};确定哪些模型组件对应于观察到的数据变量。。gastroFitObs =“[谷氨酸出现率]=谷氨酸”;估计以下参数的值:估计信息({“kmax”,“kmin”,“出租车”,“剂量”});确保参数估计值总是正的%,使用对所有参数进行日志转换。[gastroFitEst。变换= deal(交易)“日志”);将“剂量”的初始估计值设置为报告的膳食剂量量。的%剩余的初始估计值将取自%模型。gastroFitEst(4)。我nitialValue = mealDose.Amount;用初始参数估计生成模拟数据configset。StopTime=7; gastroInitSim = sbiosimulate(m1, mealDose);使用|nlinfit|拟合数据,在每次迭代中显示输出fitOptions = statset(“显示”,“通路”);[gastroFitResults, gastroFitSims] = sbiofit(m1, fitData,...gastroFitObs, gastroFitEst, [],“nlinfit”, fitOptions);
迭代SSE梯度步长的模数的模数----------------------------------------------------------- 0 43.798 1 3.1179 32.3923 0.462126 2 2.13847 1.04093 0.0510667 3 2.13751 0.000736706 0.0045159 4 2.13749 5.76665e-05 0.000866769 5 2.13749 5.87539e-06 0.000194306 6 2.13749 4.05387e-07 4.77789e-05 7 2.13749 2.0556e-08 1.31015e-05迭代终止:SSE的相对变化小于OPTIONS。TolFun
拟合数据使用fminunc
现在,使用“优化工具箱”函数估计参数fminunc.
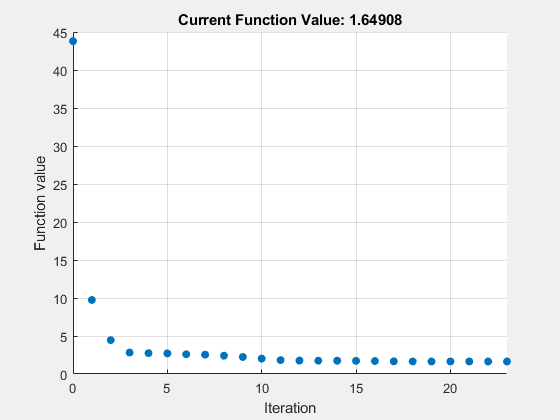
拟合数据,绘制每次迭代的目标函数fitOptions2 = optimoptions(“fminunc”,“PlotFcns”, @optimplotfval);[gastroFitResults(2), gastroFitSims(2)] = sbiofit(m1, fitData,...gastroFitObs, gastroFitEst, [],“fminunc”, fitOptions2);
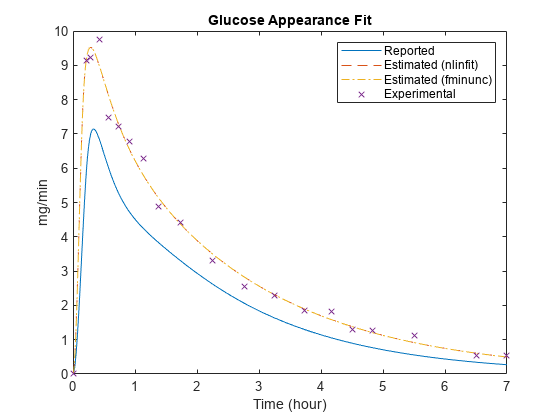
比较拟合前后的模拟结果。
胃镜= selectbyname([gastroInitSim],“Glu出现率”);图;情节(gastroSims(1)。时间,美食模拟人生(1)。数据,“- - -”,...gastroSims(2)。时间,美食模拟人生(2)。数据,“——”,...gastroSims(3)。时间,美食模拟人生(3)。数据,“-”。,...fitData。Time, fitData。GluRate,“x”);包含(的时间(小时));ylabel (毫克/分钟的);传奇(“报告”,“估计(nlinfit)”,...“估计(fminunc)”,“实验”);标题(“葡萄糖外观匹配”);
绘制相对于报告值的参数值的变化。
图;fitResults = [gastroFitResults(1).ParameterEstimates.Estimate ....gastroFitResults (2) .ParameterEstimates.Estimate];kmax、kmin和kab的初始值来自于模型。gastroFitInitValues = [get(sbioselect(m1,“名字”,“kmax”),“价值”)得到(sbioselect (m1,“名字”,“kmin”),“价值”)得到(sbioselect (m1,“名字”,“出租车”),“价值”) gastroFitEst(4)。InitialValue];relFitChange = fitResults ./ [gastroFitInitValues gastroFitInitValues] - 1;酒吧(relFitChange);Ax = gca;斧子。XTickLabel = {gastroFitEst.Name};ylabel (“估计值的相对变化”);标题(比较报告的和估计的胃肠参数值);传奇({“nlinfit”,“fminunc”},“位置”,“北”)
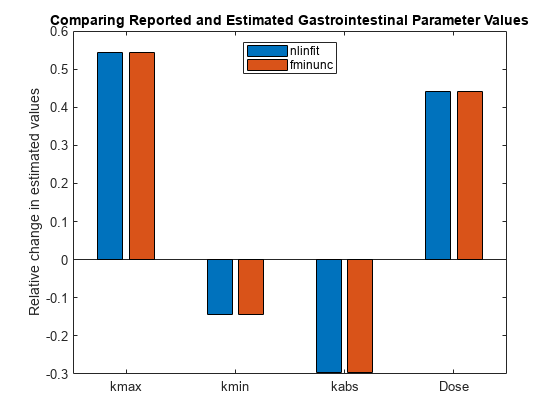
请注意,当膳食尺寸为(剂量)的值明显大于报告的值,则参数kmax比报道的要大得多,还有出租车比报告的要小。
葡萄糖利用肌肉和脂肪组织模型的拟合
肌肉和脂肪组织模型表示葡萄糖在体内是如何被利用的。这个子系统的“输入”是血浆中胰岛素的浓度(等离子体),内源性葡萄糖产生量(Glu刺激),以及葡萄糖出现的速度(谷氨酸出现率).“输出”是血浆中葡萄糖的浓度(血浆Glu Conc)和葡萄糖利用率(Glu跑龙套).
因为输入是时间的函数,所以它们需要作为强制函数来实现。的值等离子体,Glu刺激,谷氨酸出现率由重复赋值控制,调用函数对报告的实验值进行线性插值。当使用这些函数来控制物种或参数时,必须使用于设置其值的任何其他规则无效。为了便于这些规则的选择,规则Name属性包含有意义的名称。
为“输入”创建强制函数:%血浆胰岛素PlasmaInsRule = sbioselect(m1,“名字”,“等离子体合成定义”);plasmainsfortingfcn = sbioselect(m1,“名字”,“等离子体合成强迫功能”)
PlasmaInsForcingFcn = SimBiology规则数组索引:RuleType: Rule: 1 repeatedAssignment[血浆素Conc] = [picomole每升]*血浆素(时间/[1小时])
PlasmaInsRule。主动= false;PlasmaInsForcingFcn。主动=真;内源性葡萄糖产量(Glu Prod)GluProdRule = sbioselect(m1,“名字”,“Glu Prod定义”);gluprodfortingfcn = sbioselect(m1,“名字”,“Glu Prod强制功能”)
gluprodfortingfcn = SimBiology规则数组索引:规则类型:规则:1 repeatedAssignment [Glu Prod] =[毫克每分钟]*内源性葡萄糖产生(时间/[一小时])
GluProdRule。主动= false;GluProdForcingFcn。主动=真;葡萄糖出现率(Glu出现率)GluRateRule = sbioselect(m1,“名字”,“Glu出现率定义”);gluratefortingfcn = sbioselect(m1,“名字”,“Glu出现速率强制函数”)
GluRateForcingFcn = SimBiology规则数组索引:规则类型:规则:1 repeatedAssignment [Glu出现率]=[毫克每分钟]*葡萄糖出现率(时间/[一小时])
GluRateRule。主动= false;GluRateForcingFcn。主动=真;使用初始参数值进行模拟muscleInitSim = sbiosimulation (m1);确定哪些模型组件对应于观察到的数据变量。。muscleFitObs = {'[Plasma GluConc] = PlasmaGluConc',...'[Glu Util] = GluUtil'};估计以下参数的值:muscleFitEst = estimatedInfo({【血浆体积(Glu)】,“k1”,“k2”,...“Vm0”,“Vmx”,“公里”,“p2U”});确保参数估计值总是正的%,使用对所有参数进行日志转换。[muscleFitEst。变换= deal(交易)“日志”);拟合数据,在每次迭代中显示输出[muscleFitResults, muscleFitSim] = sbiofit(m1, fitData,...muscleFitObs, muscleFitEst, [],“nlinfit”, fitOptions);
迭代SSE梯度Step的Norm of Norm ----------------------------------------------------------- 0 2181.52 1 1085.53 39020.2 0.610029 2 1059.87 34535.8 0.741592 3 743.846 18217.2 0.752823 4 544.725 12620.2 0.738337 5 328.744 2879.49 0.383848 6 321.805 3860.01 0.159715 7 269.919 4675.63 0.200186 8 265.575 2548.53 0.0536833 9 263.684 2060.19 0.0294674 10 262.848 652.619 0.0294674 11 262.346 1087.79 0.00407125 12 261.897 674.32 0.00199629 14 261.523 1327.790.00115098 15 261.475 723.834 0.0025953 16 261.28 312.505 0.00210966 17 257.985 625.055 0.0336004 18 256.732 633.64 0.0167036 21 251.642 398.83 0.0677341 22 250.661 575.847 0.0124814 23 250.601 1371.22 0.00134259 24 250.509 670.542 0.00182034 25 250.341 881.41 0.00336496 26 250.205 601.85 0.0028711 27 250.136 405.799 0.0012213 29 253.329 0.02625556 31 249.718 2398.163.39399e-17迭代终止:当前步骤的相对范数小于OPTIONS。TolX
绘制相对于报告值的参数值的变化。
图;muscleFitInitValues = [get(sbioselect(m1,“名字”,“血浆体积(Glu)”),“价值”)得到(sbioselect (m1,“名字”,“k1”),“价值”)得到(sbioselect (m1,“名字”,“k2”),“价值”)得到(sbioselect (m1,“名字”,“Vm0”),“价值”)得到(sbioselect (m1,“名字”,“Vmx”),“价值”)得到(sbioselect (m1,“名字”,“公里”),“价值”)得到(sbioselect (m1,“名字”,“p2U”),“价值”));栏(muscleFitResults.ParameterEstimates。/ muscleFitInitValues - 1);Ax = gca;斧子。XTickLabel = {muscleFitEst.Name};ylabel (“估计值的相对变化”);标题(“比较报告的和估计的葡萄糖参数值”);
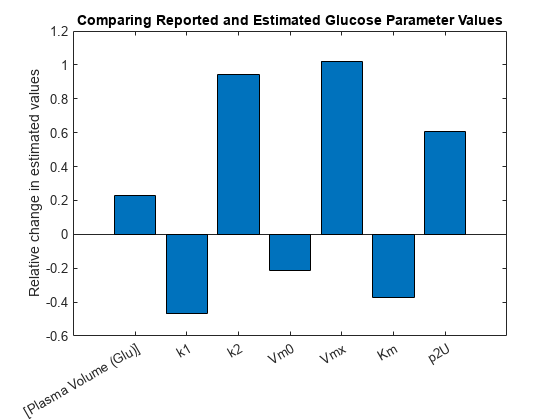
清理对模型的更改。
PlasmaInsRule。主动=真;GluProdRule。主动=真;GluRateRule。主动=真;PlasmaInsForcingFcn。主动= false;GluProdForcingFcn。主动= false;GluRateForcingFcn。主动= false;
比较拟合前后的模拟结果
muscleSims = selectbyname([muscleInitSim muscleFitSim],...{“Plasma Glu Conc”,“Glu Util”});图;情节(muscleSims(1)。时间,muscleSims (1) . data (: 1),“- - -”,...muscleSims(2)。时间,muscleSims (2) . data (: 1),“——”,...fitData。时间,fitData。PlasmaGluConc,“x”);包含(的时间(小时));ylabel (“mg / dl”);传奇(“初始(模拟)”,“估计(模拟)”,“实验”);标题(“血糖匹配”);
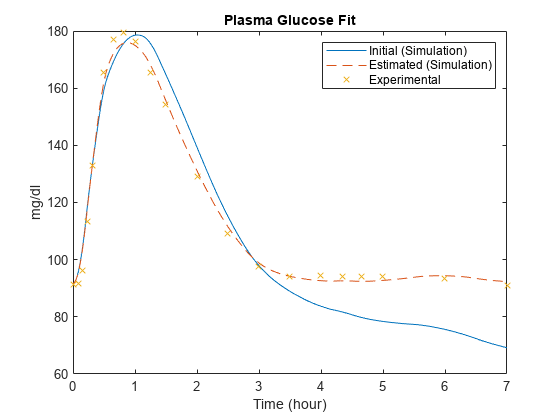
图;情节(muscleSims(1)。时间,muscleSims (1) . data (:, 2),“- - -”,...muscleSims(2)。时间,muscleSims (2) . data (:, 2),“——”,...fitData。时间,fitData。GluUtil,“x”);包含(的时间(小时));ylabel (毫克/分钟的);传奇(“初始(模拟)”,“估计(模拟)”,“实验”);标题(“葡萄糖利用适合”);
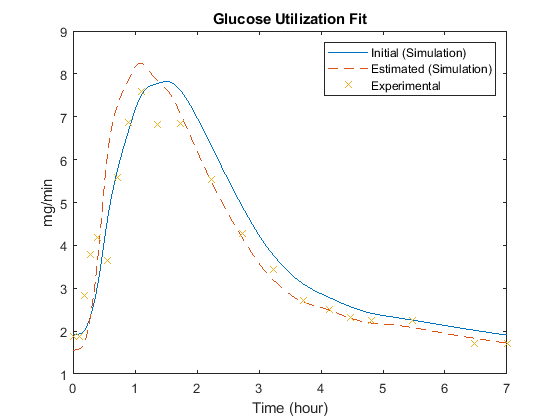
注意,显著增加一些参数,例如Vmx可以更好地拟合晚期血糖浓度。
清理
恢复警告设置。
警告(warnSettings);
结论
SimBiology包含几个特性,可以促进葡萄糖-胰岛素系统复杂模型的实现和模拟。反应、事件和规则提供了一种描述系统动态的自然方式。单位转换可以方便地以单位指定物种和参数,并保证模型的尺寸一致性。剂量对象是描述模型重复输入的一种简单方法,例如本例中的每日用餐计划。SimBiology还为模拟和参数估计等分析任务提供了内置支持。金宝app

