RNA序列二级结构的预测与可视化
这个例子演示了如何使用rnafold.和rnaplot用于预测和绘制RNA序列的二级结构的功能。
介绍
RNA在细胞中扮演着重要的角色,既是遗传信息载体(mRNA),也是功能元件(tRNA, rRNA)。由于RNA序列的功能很大程度上与其结构有关,因此从其序列预测RNA的结构变得越来越重要。由于碱基配对和碱基堆积是对折叠的自由能贡献的主要部分,因此,良好的二级结构估计不仅有助于解释RNA分子的功能和反应性,而且有助于分析RNA分子的三级结构。
利用最近邻热力学模型预测RNA二级结构
RNA序列的二级结构由其基部之间的相互作用确定,包括氢键和基部堆叠。RNA二次结构预测的许多方法之一使用最近邻模型,并最大限度地减少与RNA结构相关的总自由能量。通过从基对堆叠,发夹,凸起,内部环和多分支环路求和各个能量贡献来估算最小自由能量。这些元素的能量贡献是序列和长度依赖性的,并且已经通过实验确定[1]。这rnafold.功能使用最近邻的热力学模型来预测RNA序列的最小自由能二次结构。更具体地,该算法实施在rnafold.利用动态规划方法计算所有可能的初等子结构的能量贡献,然后通过考虑总自由能最小的初等子结构的组合来预测二级结构。在此计算中,不考虑同轴叠加螺旋的贡献,也不考虑伪结(非嵌套结构单元)的形成。
转移RNA苯丙氨酸的二次结构
trna是一种小分子(73-93个核苷酸),在转译过程中将特定的氨基酸转移到核糖体位置的生长多肽链上。虽然每个氨基酸类型至少存在一个tRNA分子,但不同tRNA类型之间的二级和三级结构都很保守,这很可能是由于需要与核糖体保持可靠的相互作用。我们考虑下面的tRNA-Phe序列酿酒酵母并预测使用该功能的最小自由能二次结构rnafold..
% ===用括号表示预测二级结构phe_seq ='gcggauuuagcucaguugggagagagcgccagacugaagaucuggagguguguuguucgauccacagaauucgcacca';phe_str = rnafold (phe_seq)
phe_str = '(((((((..((((........)))).(((((.......))))).....(((((.......))))))))))))....'
在括号表示法中,每个点代表一个未配对的基数,而一对嵌套相同的前后括号代表一个基数对。利用该函数可以绘制RNA二级结构的替代表示rnaplot.例如,上面预测的结构可以显示为根树,其中叶节点对应于未配对的残差和内部节点(除根之外)对应于基对。您可以通过单击相应的节点显示每个残差的位置和类型。
% ===图RNA二级结构为树rnaplot(phe_str,“>”phe_seq,“格式”,“树”);

TRNA二级结构通常在图中表示并类似于三叶草叶。它显示出四个基部配对的杆(或“臂”)和三个环。四个茎中的每一个已被广泛研究和表征:受体阀杆(位置1-7和66-72),D-阀杆(位置10-13和22-25),抗ocon杆(位置27-31和39-43)和T-茎(位置49-53和61-65)。我们可以将TRNA二级结构绘制为二维图,其中每残基通过点和骨架鉴定,并且氢键表示为点之间的线。茎由连续的碱基配对残基(蓝点)组成,而环绕由未配对的残基(红色点)形成。
%===使用点图表示绘制辅助结构rnaplot(phe_str,“>”phe_seq,“格式”,'点');文本(500、200、“T-stem”);文本(100、600、'反恋茎');文本(550、650、“D-stem干”);文本(700、400、'受体茎');

虽然所有的茎对分子的三维折叠和成功地与核糖体和tRNA合成酶相互作用都很重要,但受体茎和反密码子茎特别有趣,因为它们包括附着位点和反密码子三联体。附着位点(位置74-76)位于RNA链的3'端,由所有氨基酸受体茎中的C-C-A序列组成。反密码子三联体由3个碱基组成,它们与信使RNA中的互补密码子配对。在phetrna中,反密码子序列A-A-G(位置34-36)与mRNA密码子U-U-C配对,编码氨基酸苯丙氨酸。我们可以重新绘制结构,并突出这些区域的受体干和反密码子干使用选择属性:
aag_pos = 34:36;cca_pos = 74:76;rnaplot(phe_str,'顺序'phe_seq,“格式”,'图表',...“选择”(aag_pos cca_pos]);

通过将结构显示为图绘图,更好地理解序列的分离成四个单独的杆。每个残留物在横坐标和半椭圆线上表示,彼此配对的碱基。通过不存在交叉线来反映次要结构中的缺失。这是在TRNA二级结构中的预期和预期,因为使用的动态编程方法不允许伪动力。
rnaplot(phe_str,'顺序'phe_seq,“格式”,'图形');

类似的观察结果可以通过将二级结构显示为一个圆来绘制,其中每个基底都由任意大小的圆周长上的一个点表示,相互配对的基底用线连接。这些线在视觉上被聚集成四个不同的组,由未配对残基的延伸分开。我们可以用H.Unpaired,把手返回了colorby物业设为状态.
[ha, H] = rnaplot(phe_str,'顺序'phe_seq,“格式”,'圆圈',...'colorby',“状态”);H.Unpaired.Visible ='离开';传说从;

正如您所看到的,所以rnaplot功能包括MATLAB®结构H由可用于改变各种残差子集的方面属性的手柄组成。例如,如果您使用该方法设置颜色方案colorby物业设为渣,这些点是根据残留类型上色的,你可以使用适当的手柄改变它们的属性。
[ha, H] = rnaplot(phe_str,'顺序'phe_seq,“格式”,'圆圈','colorby','残留')
公顷=轴(Bioinfo: rnaplot:圆)属性:XLim: [1] YLim: 1.1000 [1] XScale:“线性”YScale:“线性”GridLineStyle:“-”位置:[0.1125 0.1100 0.6709 0.8150)单位:“正常化”的使用得到显示所有属性H =结构体字段:答:[1 x1线]C: [1 x1线]G: [1 x1线]U: [1 x1线]选择:(0 x1线)

H.G.Color = [0.5 0.5 0.5];H.G.Marker =.‘*’;H.C.Color = [0.5 0.5 0.5];H.C.Marker =“+”;

苯基丙氨酸转移RNA的保存
尽管tRNAs分子的一级序列存在一些差异,但其二级结构模式在三个系统发育域中都很保守。考虑一个具有代表性的生物的每个系统发育域的tRNA-Phe结构:酿酒酵母真核生物的Haloarcula Marismortiui.为古生菌,和热嗜热疗法对于细菌。然后使用山绘图表示预测并绘制其二级结构。
酵母=“GCGGACUUAGCUCAGUUGGGAGAGCGCCAGACUGAAGAUCUGGAGGUCCUGUGUUCGAUCCACAGAGUUCGCACCA”;豪迈='gccgcuuagcacagacugggagagagcacucgacaagaucgagcuguccccguucaaauccgggaggcggcacca';theth =“GCCGAGGUAGCUCAGUUGGUAGAGCAUGCGACUGAAAAUCGCAGUGUCGGCGGUUCGAUUCCGCCCCUCGGCACCA”;yeast_str = rnafold(酵母);theth_str = rnafold (theth);halma_str = rnafold(豪迈);h1 = rnaplot (yeast_str,'顺序', 酵母,“格式”,“山”);标题(h1,'trna-phe saccharomyces cerevisiae');传说隐藏;h2 = rnaplot (halma_str,'顺序'豪迈,“格式”,“山”);标题(h2,'trna-phe haloarcula marismortui');传说隐藏;h3 = rnaplot(theth_str,'顺序'theth,“格式”,“山”);标题(h3,“tRNA-Phe栖热菌属酸奶的);传说隐藏;



结果结构之间的相似性是惊人的,唯一的区别是d环上多了一个残差Haloarcula Marismortiui.,显示在山地块的第一个平坦的斜坡上。
G-U摆动碱基对
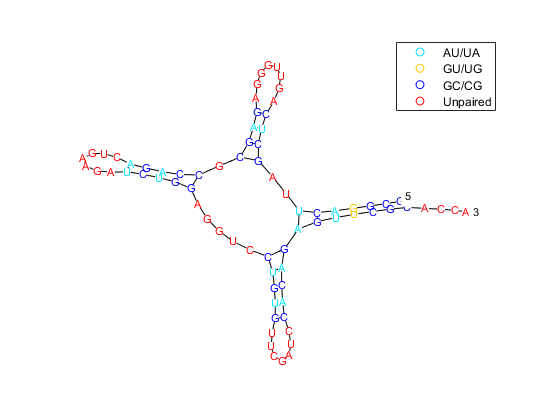
除了沃森-克里克碱基对(A-U, G-C)外,几乎每一类功能性RNA都有G-U摇摆碱基对。G-U对具有一系列独特的化学、结构和构象性质:它们对金属离子具有很高的亲和力,在热力学上几乎与沃森-克里克碱基对一样稳定,并且在不同的环境中具有构象灵活性。tRNA受体螺旋第三位的摆动对在几乎所有的生物中都是高度保守的。这种保守性表明G-U对具有其他配对难以复制的独特特征。根据碱基对的类型给碱基对上色,可以在二级结构图上观察碱基对的类型分布。
rnaplot (yeast_str'顺序', 酵母,“格式”,'图表','colorby',“对”);
参考文献
[1]马修斯,D.,Sabina,J.,Zuker,M.和特纳,D。“热力学参数的扩展序列依赖性提高了RNA二级结构的预测”,分子生物学,288(5):911-40,1999。

