基因表达分析
这个例子演示了使用神经网络在面包酵母中寻找基因表达谱的模式。
问题:分析贝克酵母(Saccharomyces Cerevisiae)的基因表达
目的是对酿酒酵母(Saccharomyces cerevisiae)的基因表达有一定的了解,这种酵母通常被称为面包酵母或啤酒酵母。它是一种用来烤面包和用葡萄发酵葡萄酒的真菌。
当酵母被引入到富含葡萄糖的培养基中时,它可以将葡萄糖转化为乙醇。最初,酵母通过一种称为“发酵”的代谢过程将葡萄糖转化为乙醇。然而,一旦葡萄糖供应耗尽,酵母就会从葡萄糖的厌氧发酵转变为乙醇的有氧呼吸。这个过程被称为二auxic shift。这一过程是相当有趣的,因为它伴随着基因表达的重大变化。
该实例使用DNA微阵列数据来研究酿酒酵母中几乎所有基因在二氧移期间的时间基因表达。
您需要Bioinformatics Toolbox™来运行此示例。
如果~ nnDependency。bioInfoAvailable errordlg (“这个例子需要生物信息学工具箱。”);返回;结束
的数据
本例使用来自DeRisi、JL、Iyer、VR、Brown、PO的数据。“在基因组尺度上探索基因表达的代谢和遗传控制。”科学,1997年10月24日;278(5338):680-6。PMID: 9381177
完整的数据集可以从基因表达Omnibus网站下载:https://www.yeastgenome.org
首先将数据加载到MATLAB®中。
负载yeastdata.mat
基因表达水平的测量在7个时间点在二氧移。的变量次包含在实验中测量表达水平的时间。的变量基因包含已测量表达水平的基因的名称。的变量yeastvalues包含实验中七个时间步的“VALUE”数据或LOG_RAT2N_MEAN,或CH2DN_MEAN与CH1DN_MEAN比值的log2。
为了了解您可以使用的数据的大小元素个数(基因)来显示数据集中有多少基因。
元素个数(基因)
Ans = 6400
基因是一个细胞阵列的基因名称。您可以使用MATLAB单元格数组索引访问条目:
基因{15}
ans = 'YAL054C'
这表示变量的第15行yeastvalues包含ORF的表达式级别YAL054C.
筛选基因
数据集相当大,很多信息对应的基因在实验中没有显示出任何有趣的变化。为了更容易找到感兴趣的基因,首先要做的是通过删除表达谱不显示任何感兴趣的基因来减少数据集的大小。共有6400个表达式配置文件。你可以使用一些技术将其减少到包含最重要基因的子集。
如果你浏览基因列表,你会看到几个标记为“空”的点。这些是数组上的空点,虽然它们可能有与之关联的数据,但对于本例来说,您可以将这些点视为噪声。这些点可以用比较字符串函数,并使用索引命令从数据集中删除。
空点= strcmp(“空”,基因);yeastvalues(emptySpots,:) = [];基因(emptySpots) = [];元素个数(基因)
Ans = 6314
在yeastvalues数据中,您还将看到有几个地方的表达式级别被标记为NaN。这表明在特定的时间步骤中没有收集到该点的数据。处理这些缺失值的一种方法是使用特定基因随时间变化的数据的平均值或中位数来估算它们。这个例子使用了一种不那么严格的方法,即简单地丢弃没有测量一个或多个表达水平的任何基因的数据。
这个函数isnan用于识别缺失数据的基因,索引命令用于删除缺失数据的基因。
nanIndices = any(isnan(yeastvalues),2);yeastvalues(nanIndices,:) = [];基因(nanIndices) = [];元素个数(基因)
Ans = 6276
如果您要绘制所有剩余概要文件的表达式概要文件,您将看到大多数概要文件是平坦的,与其他概要文件没有显著不同。这一平坦的数据显然是有用的,因为它表明与这些剖面相关的基因不受二适性移位的显著影响;然而,在这个例子中,你感兴趣的是伴随二氧移的表达有很大变化的基因。您可以使用生物信息学工具箱™中的过滤功能来删除具有各种类型的配置文件的基因,这些配置文件不能提供有关受代谢变化影响的基因的有用信息。
您可以使用genevarfilter函数过滤出随时间变化的小变异基因。该函数返回一个与可变基因大小相同的逻辑数组,其中1对应于方差大于第10个百分位的酵母值行,0对应于低于阈值的酵母值行。
掩码= genevarfilter(yeastvalues);%使用掩码作为值的索引,以删除过滤的基因。Yeastvalues = Yeastvalues (mask,:);基因=基因(面具);元素个数(基因)
Ans = 5648
这个函数genelowvalfilter去除绝对表达值非常低的基因。注意,基因过滤函数还可以自动计算过滤后的数据和名称。
[掩膜,酵母值,基因]=...genelowvalfilter (yeastvalues基因,“absval”log2 (3));元素个数(基因)
Ans = 822
使用geneentropyfilter去除谱图熵低的基因:
[掩膜,酵母值,基因]=...geneentropyfilter (yeastvalues基因,“prctile”15);元素个数(基因)
Ans = 614
主成分分析
现在你已经有了一个可管理的基因列表,你可以寻找档案之间的关系。
将数据的标准差和均值归一化可以使网络在其值范围内将每个输入视为同等重要。
主成分分析(PCA)是一种有用的技术,可用于降低大型数据集的维数,如微阵列分析。该技术隔离了数据集的主要组件,消除了那些对数据集中的变化贡献最小的组件。
可以使用这两个设置变量进行应用mapstd而且processpca对新数据保持一致。
[x,std_settings] = mapstd(yeastvalues');规范化数据[x,pca_settings] = processpca(x,0.15);% PCA
输入向量首先被归一化,使用mapstd,使它们的均值为零,方差为单位。processpca是实现PCA算法的函数。第二个参数传递给processpca是0.15。这意味着processpca消除了对数据集中总变化贡献小于15%的主成分。的变量个人电脑现在包含了酵母值数据的主要组成部分。
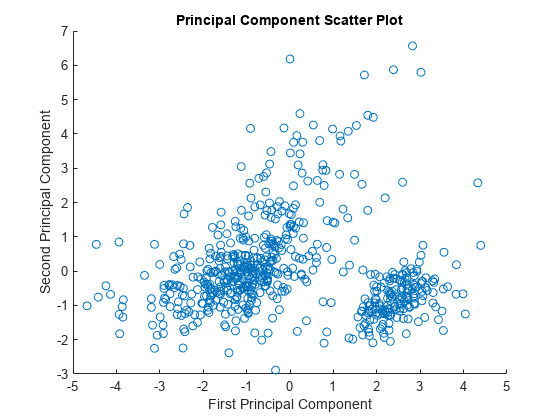
控件可以可视化主组件散射函数。
图散射(x (1:), (2,:));包含(“第一主成分”);ylabel (“第二主成分”);标题(“主成分散点图”);
聚类分析:自组织地图
现在可以使用自组织映射(SOM)聚类算法对主组件进行聚类。
的selforgmap函数创建一个自组织映射网络,然后可以使用火车函数。
输入大小为0,因为网络还没有配置为与输入数据匹配。当网络被训练时,就会发生这种情况。
Net = selforgmap([5 3]);视图(净)
现在可以训练网络了。
神经网络训练工具显示了正在训练的网络和用于训练它的算法。它还显示训练期间的训练状态,停止训练的标准将以绿色突出显示。
底部的按钮可以打开有用的图,可以在训练期间和训练结束后打开。算法名称和绘图按钮旁边的链接打开了关于这些主题的文档。
Net = train(Net,x);

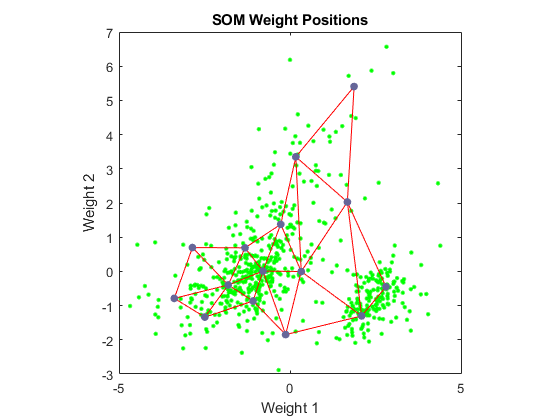
使用plotsompos在数据的前两个维度的散点图上显示网络。
图plotsompos(净,x);
您可以使用SOM为数据集中的每个点找到最近的节点来分配集群。
Y = net(x);cluster_indexes = vec2ind(y);
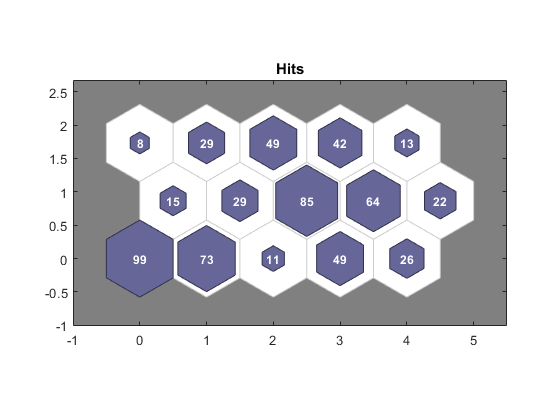
使用plotsomhits看看有多少向量被分配给了地图上的每个神经元。
图plotsomhits(净,x);
您还可以使用其他聚类算法,如分层聚类和K-means,在统计和机器学习工具箱™中可用于聚类分析。
术语表
羊痘疮-开放阅读框(ORF)是基因序列的一部分,它包含一个碱基序列,不受停止序列的干扰,可能编码蛋白质。

