利用交互式NDVI阈值法识别植被区域
这个例子展示了如何通过归一化植被指数差(NDVI)图的交互阈值来识别高光谱图像中的植被区域类型。高光谱数据集的NDVI图反映了高光谱数据不同区域的植被密度。利用高光谱数据立方体中的近红外(NIR)和可见红色(R)光谱波段图像计算NDVI值。
像素的NDVI值为-1 ~ 1之间的标量。植被健康或茂密地区的像素反射更多的近红外光,导致NDVI值较高。植被不健康或土地贫瘠地区的像素吸收更多的近红外光,导致NDVI值较低或为负。根据NDVI值,可以将一个地区的植被识别为茂密植被、中等植被、稀疏植被,或没有植被.以下是每种类型区域的典型NDVI值范围:
密集植被-大于或等于0.6
中度植被-在[0.4,0.6)范围内
稀疏植被-范围[0.2 0.4)
无植被-低于0.2
您可以通过使用NDVI值执行阈值分割所需的植被区域。在本例中,您将交互式地选择和更改阈值,以基于NDVI值识别高光谱数据立方体中的不同植被区域。
读取高光谱数据
从ENVI格式文件读取高光谱数据到工作空间。本例使用来自Pavia数据集的数据样本,其中包含植被和贫瘠地区。
超立方(“paviaU.dat”,“paviaU.hdr”);
计算归一化植被指数
方法计算数据立方体中每个像素的NDVI值归一化植被指数函数。该函数输出一个二维图像,其中每个像素的值是高光谱数据立方体中相应像素的NDVI值。
ndviImg = ndvi(hcube);
利用阈值法识别植被区域
利用多级阈值法识别高光谱数据中的不同区域。定义一个标签矩阵,根据指定的阈值为像素分配标签值。用户可以根据NDVI计算值设置阈值。
标签的值
1—指定阈值为0.6,找出NDVI值大于或等于该阈值的像素点。这些是密集的植被像素。标签的值
2—下限阈值和上限阈值分别为0.4和0.6。找出NDVI值大于等于0.4小于0.6的像素。这些是中等植被像素。标签的值
3.—下限阈值和上限阈值分别为0.2和0.4。找出NDVI值大于等于0.2小于0.4的像素。这些是稀疏的植被像素。标签的值
4—指定阈值为0.2,找出NDVI值小于阈值的像素点。这些不是植被像素。
L = 0 (size(ndviImg));L(ndviImg >= 0.6) = 1;L(ndviImg >= 0.4 & ndviImg < 0.6) = 2;L(ndviImg >= 0.2 & ndviImg < 0.4) = 3;L(ndviImg < 0.2) = 4;
方法从原始数据立方体中估计对比度拉伸的RGB图像彩色化函数。
rgbImg = colorize(hcube,“方法”,“rgb”,“ContrastStretching”,真正的);
定义一个colormap以不同的颜色显示标签矩阵中的每个值。将标签矩阵覆盖在RGB图像上。
Cmap = [0 1 0;0 0 1;1 10 0;10 0 0];overlayImg = labeloverlay(rgbImg,L,“Colormap”,提出);
创建NDVI阈值化交互界面
方法来创建一个交互式界面,首先要创建一个图形窗口uifigure函数。然后在图形窗口中添加两个面板,用于并排显示输入图像和叠加图像。
H = uifigure(“名字”,“交互式NDVI阈值化”,“位置”, 200, 1000, 700);viewPanel1 = uipanel(h,“位置”,[2 220 400 450],“标题”,输入图像的);ax1 = axes(viewPanel1);图像(rgbImg,“父”viewPanel2 = uipanel(h,“位置”,[400 220 400 450],“标题”,“输入图像中的植被区域类型”);ax2 = axes(viewPanel2);图像(overlayImg,“父”, ax2)
用每个标签及其相关植被密度的颜色注释图形窗口。密集植被的颜色图值为绿色,中等植被为蓝色,稀疏植被为黄色,无植被为红色。
注释(h,“矩形”,[0.82 0.82 0.03 0.03],“颜色”,[0 1 0],“FaceColor”,[0 1 0]);注释(h,“矩形”,[0.82 0.77 0.03 0.03],“颜色”,[0 0 1],“FaceColor”,[0 0 1]);注释(h,“矩形”,[0.82 0.72 0.03 0.03],“颜色”,[1 10 0],“FaceColor”,[1 1 0]);注释(h,“矩形”,[0.82 0.67 0.03 0.03],“颜色”,[10 0 0],“FaceColor”,[10 0 0]);注释(h,“文本框”,[0.85 0.80 0.9 0.05],“EdgeColor”,“没有”,“字符串”,茂密的植被的);注释(h,“文本框”,[0.85 0.75 0.9 0.05],“EdgeColor”,“没有”,“字符串”,“温和的植物”);注释(h,“文本框”,[0.85 0.70 0.9 0.05],“EdgeColor”,“没有”,“字符串”,“稀疏植被”);注释(h,“文本框”,[0.85 0.65 0.9 0.05],“EdgeColor”,“没有”,“字符串”,“没有植被”);
创建用于交互更改阈值的滑块。使用uislider函数添加一个调节最小阈值的滑块和一个调节最大阈值的滑块。
slidePanel1 = uipanel(h,“位置”(400120400、70),“标题”,“最小阈值”);minsld = uislider(slidePanel1,“位置”(40350 3),“价值”, 1“限制”[1],“MajorTicks”, 1:0.4:1);slidePanel2 = uipanel(h,“位置”(400、30400、70),“标题”,“最大阈值”);maxsld = uislider(slidePanel2,“位置”(35350 3),“价值”,1,“限制”[1],“MajorTicks”, 1:0.4:1);
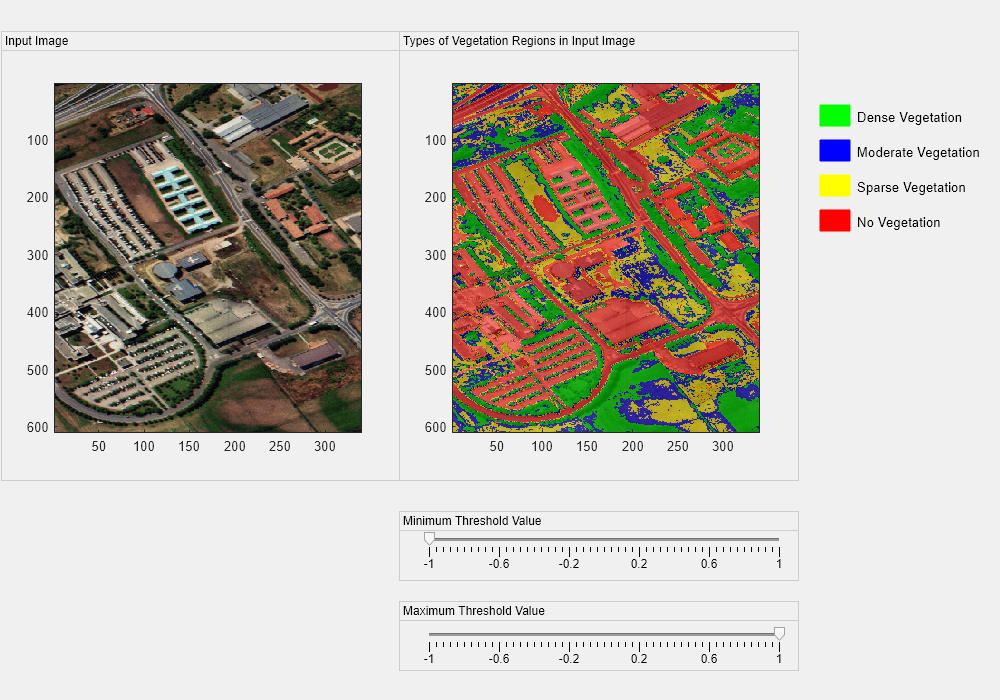
交互更改阈值
使用函数ndviThreshold更改最小和最大阈值限制。当您移动滑块拇指并释放鼠标按钮时,ValueChangedFcn回调函数更新滑块值,并将滑块值设置为新的阈值。你必须打电话给ndviThreshold最小阈值滑块和最大阈值滑块分别作用。通过调整滑块更改阈值限制。这使您能够在指定的阈值限制内检查植被区域的类型。
minsld。ValueChangedFcn = @(es,ed) ndviThreshold(minsld,maxsld,ndviImg,rgbImg,ax2,cmap);maxsld。ValueChangedFcn = @(es,ed) ndviThreshold(minsld,maxsld,ndviImg,rgbImg,ax2,cmap);
的ndviThreshold函数使用更新的阈值生成一个新的标签矩阵,并动态更新图窗口中覆盖的图像。
创建回调函数
创建回调函数以交互方式更改阈值限制并动态更新结果。
函数ndviThreshold(minsld,maxsld,ndviImg,rgbImg,ax2,cmap) L = 0 (size(ndviImg));minth = round(minsld.Value,2);maxth = round(maxsld.Value,2);如果Minth >最大th错误('最小阈值必须小于最大阈值')结束如果Minth >= 0.6%标记1为茂密植被L(ndviImg >= minth & ndviImg <= maxth) = 1;overlayImg = labeloverlay(rgbImg,L,“Colormap”,提出);elseifMinth >= 0.4 && Minth < 0.6%标记1为茂密植被%标签2为中度植被如果maxth >= 0.6 L(ndviImg >= minth & ndviImg < 0.6) = 2;L(ndviImg >= 0.6 & ndviImg <= maxth) = 1;其他的L(ndviImg >= minth & ndviImg < maxth) = 2;结束overlayImg = labeloverlay(rgbImg,L,“Colormap”,提出);elseifMinth >= 0.2 && Minth <0.4%标记1为茂密植被%标签2为中度植被%标记3为稀疏植被如果maxth < 0.4 L(ndviImg >= minth & ndviImg <= maxth) = 3;elseif(>= minth & ndviImg < 0.4) = 3;L(ndviImg >= 0.4 & ndviImg <= maxth) = 2;elseifmaxth >= 0.6 L(ndviImg >= minth & ndviImg < 0.4) = 3;L(ndviImg >= 0.4 & ndviImg < 0.6) = 2;L(ndviImg >= 0.6 & ndviImg <= maxth) = 1;结束overlayImg = labeloverlay(rgbImg,L,“Colormap”,提出);elseifMinth < 0.2%标记1为茂密植被%标签2为中度植被%标记3为稀疏植被%标签4表示无植被L(ndviImg >= minth & ndviImg < 0.2) = 4;如果(ndviImg >= 0.6 & ndviImg <= maxth) = 1;L(ndviImg >= 0.4 & ndviImg < 0.6) = 2;L(ndviImg >= 0.2 & ndviImg < 0.4) = 3;elseif(ndviImg >=0.4 && ndviImg <= maxth) = 2;L(ndviImg >= 0.2 & ndviImg < 0.4) = 3;elseif(ndviImg >= maxth & ndviImg < 0.4) = 3;结束overlayImg = labeloverlay(rgbImg,L,“Colormap”,提出);elseifMinth == maxth如果maxth < 0.2 L(ndviImg == minth) = 4;elseifmaxth >=0.4 && maxth < 0.6 L(ndviImg == maxth) = 2;elseifmaxth >=0.2 && maxth < 0.4 L(ndviImg == maxth) = 3;elseif>= 0.6 L(ndviImg == maxth) = 1;结束overlayImg = labeloverlay(rgbImg,L,“Colormap”,提出);结束显示叠加图像。图像(overlayImg,“父”, ax2);%存储更新的标签图像minsld。使用rData = L; maxsld.UserData = L;结束
参考文献
J.W.劳斯,R.H.哈斯,J.A.谢尔,D.W.迪尔林。"用ERTS监测大平原的植被系统"第三届地球资源技术卫星1号研讨会论文集, 1:309-17。Greenbelt, NASA SP-351,华盛顿特区,1973年。
[2] Haboudane, D. <预测作物冠层绿色LAI的高光谱植被指数和新算法:在精确农业背景下的建模和验证>。环境遥感90年,没有。3(2004年4月15日):337-52。https://doi.org/10.1016/j.rse.2003.12.013。
另请参阅
超立方体|彩色化|uifigure|uipanel|uislider|labeloverlay
