主要内容
sbiompgsa
执行多参数全局灵敏度分析(需要统计和机器学习工具箱)
句法
描述
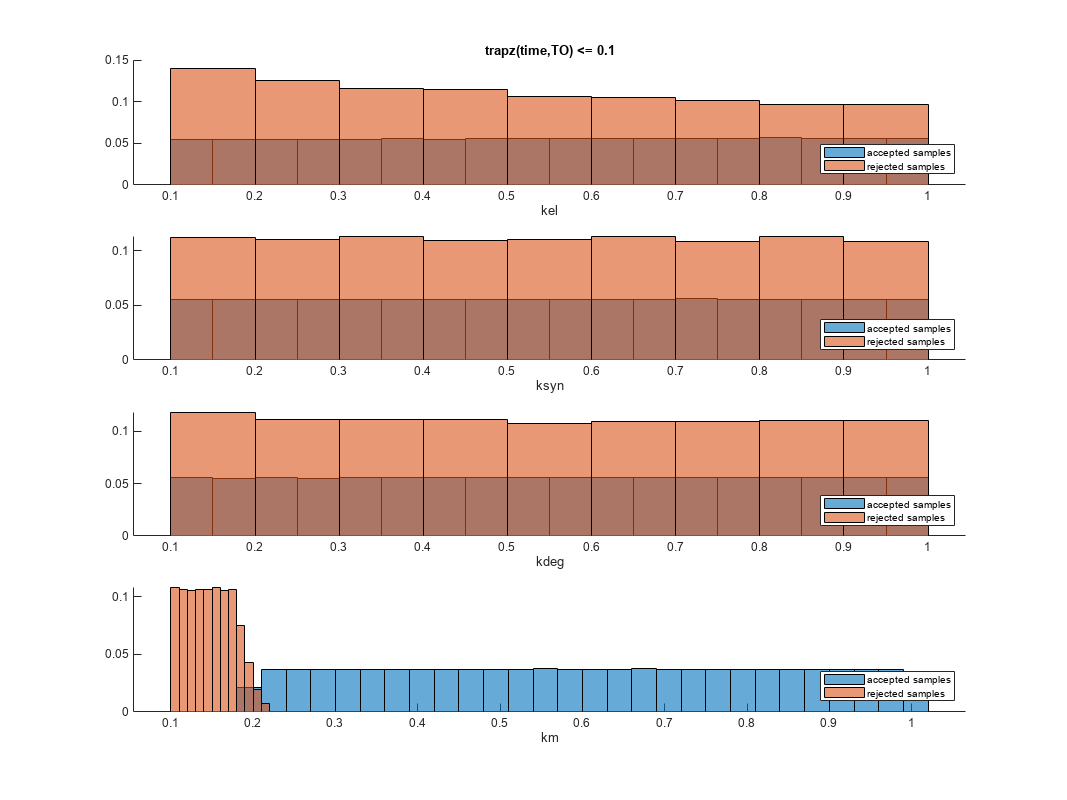
mpgsaresults.= sbiompgsa (Modelobj.那样本那分类器)样本执行多次全局敏感性分析分类器。
mpgsaresults.= sbiompgsa (simdata那样本那分类器)simdata执行多次全局敏感性分析分类器。
mpgsaresults.= sbiompgsa (___那名称,值)
例子
输入参数
输出参数
更多关于
参考
[1]Tiemann, Christian A., Joep Vanlier, Maaike H. Oosterveer, Albert K. Groen, Peter A. J. Hilbers, Natal A. W. van Riel。“参数轨迹分析以确定药物干预的治疗效果。”斯科特·马克尔编辑。PLoS计算生物学9,不。8(2013年8月1日):E1003166。https://doi.org/10.1371/journal.pcbi.1003166。
介绍了R2020a