主要内容
nbintest
小样本量计数资料的非配对假设检验
描述
测试= nbintest (X,Y)
短读计数采用负二项分布建模。
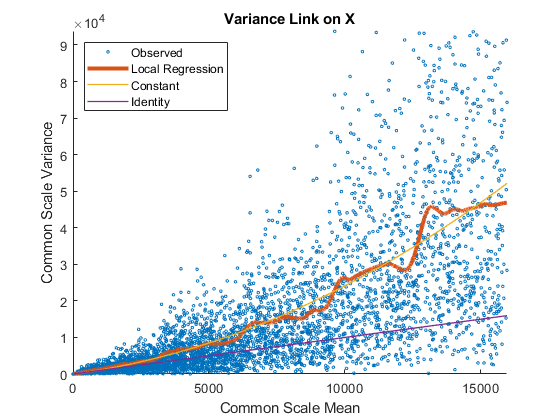
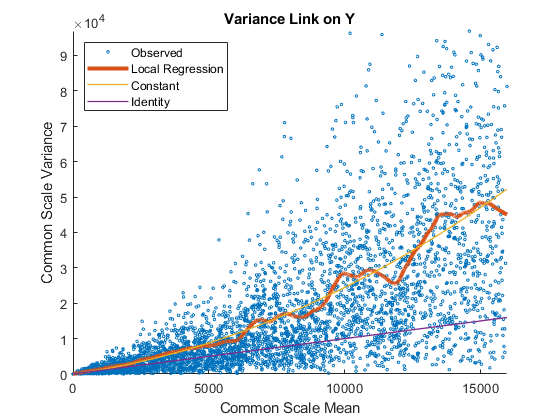
每一行数据的方差和平均值通过所有行上的回归函数相连。
X和Y必须有相同的行数和至少2列,但不一定相同的列数。行X和Y对应变量、特征或基因,如不同基因的基因表达量。列通常是时间点或病人。
测试是一个NegativeBinomialTest对象中存储的双面p值pValue财产。
当您想用小样本量(最多10个数量级)对短读计数数据(来自高通量分析,如RNA-Seq或ChIP-Seq)执行非配对假设检验时,请使用此功能。例如,使用这个函数来判断在给定的基因中,两种条件下读取计数的观察差异是否显著。
测试= nbintest (X,Y,名称,值)名称,值对参数。
请注意
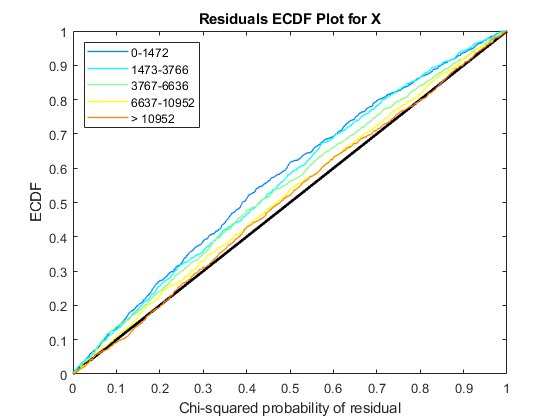
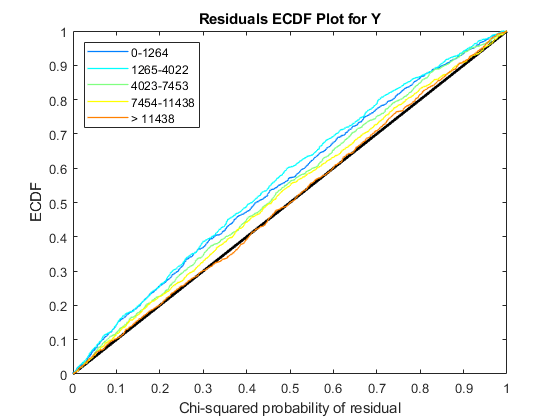
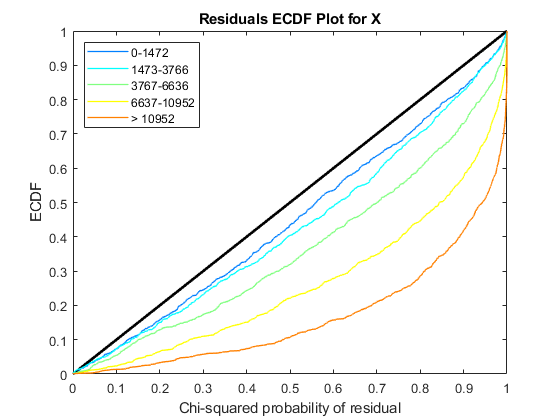
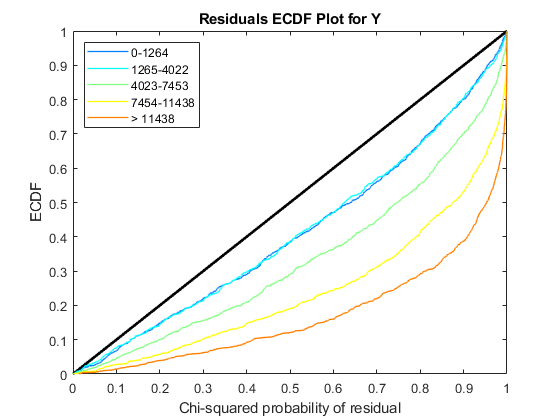
的诊断图NegativeBinomialTest返回的对象nbintest在解释p值之前。这些图允许您查看模型假设是否正确,以及所使用的方差链接是否适合于数据。
例子
输入参数
输出参数
参考文献
[1] Anders, S.和Huber, W.(2010)。序列计数数据的差异表达分析。基因组生物学、11 (10):R106。
[2] Robinson, m.d.和Smyth, G.K.(2008)。负二项离散度的小样本估计,及其在SAGE数据中的应用。生物统计学,9:321 - 332。
[3] Marioni, j.c., Mason, c.e., Mane, s.m., Stephens, M.,和Gilad, Y.(2008)。RNA-seq:技术重现性的评估和与基因表达阵列的比较。基因组研究,16:1509 - 1517。
介绍了R2014b