从RNA-Seq数据中识别差异表达基因
这个例子展示了如何使用负二项模型检测差异表达基因的RNA-Seq数据。
介绍
典型的RNA-Seq数据差异表达分析包括对原始计数进行归一化,并进行统计检验以拒绝或接受两组样本基因表达无显著差异的无效假设。这个例子展示了如何检查原始计数数据的基本统计,如何确定计数规范化的大小因素,以及如何使用负二项模型推断最差异表达的基因。
本例的数据集由Brooks等人[1]在实验中获得的RNA-Seq数据组成。作者研究了帕西拉的siRNA敲除效应,帕西拉是一个已知的在剪接调节中发挥重要作用的基因黑腹果蝇.数据集包括2个对照(未处理)样本的生物复制和2个敲除(处理)样本的生物复制。
检查读取计数表以进行基因组特征
该分析RNA-SEQ数据的起点是计数矩阵,其中行对应于感兴趣的基因组特征,该列对应于给定的样本,并且该值表示映射到给定样本中的每个特征的读取的数量。
包括文件pasilla_count_nomm.mat.包含两个表,其中包含每个所考虑样本的基因水平和外显子水平的计数矩阵。您可以使用函数获得类似的矩阵featurecount.
负载pasilla_count_nomm.mat.
%预览基因的读取计数表基因计数表(1:10,:)
ans = 10 x6表ID引用untreated3 untreated4 treated2 treated3 _______________ _________ __________ __________ ________ ________ {' FBgn0000003 '}{“3 r”}0 1 1 2{‘FBgn0000008} {2 r的}142 117 138 132{‘FBgn0000014}{“3 r”}20 12 10 19{‘FBgn0000015}{“3 r”}2 4 0 1{‘FBgn0000017} {' 3 l '} 6591 5127 4809 6027{‘FBgn0000018} {2 l的}469 530 492574 {'FBgn0000024'} {'3R'} 5 6 10 8 {'FBgn0000028'} {'X'} 002 1 {'FBgn0000032'} {'3R'} 1160 1143 1138 1415 {'FBgn0000036'} {'3R'} 000 1
注意,当计数不进行汇总时,单个特征(在本例中为外显子)被报告为元特征赋值(在本例中为基因),然后是起始和终止位置。
%预览外显子的读计数表外来电压(1:10,:)
2.治疗2治疗2治疗2 3)治疗2 2)治疗2 2 2)治疗2 2 2 2 2治疗3 3 3.UUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUUU3"2648220"2648518"{3R}2 2 2 6 6 2 2 2 6 6 6 6 2 2 2 2 2 2 2 2 2 5 5 5 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 5 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0{'3L'}1807 1572 1557 1702
您可以通过创建逻辑向量对样本进行注释和分组,如下所示:
Samples = GenecountTable(:,3:结束).properties.variablenames;未经治疗= strncmp(样品,“未经治疗”,长度(“未经治疗”)处理= strncmp(样本,“治疗”,长度(“治疗”))
未处理= 1x4逻辑阵列1 1 0 0处理= 1x4逻辑阵列0 0 1 1
绘制特性分配图
包含的文件还包含一个表geneSummaryTable已分配和未分配的SAM条目摘要。可以绘制的基本分布计算结果通过考虑读取的数量被分配到给定的基因组特性(外显子或基因对于这个示例),以及读取的数量未赋值的(即没有重叠特性)或模棱两可(即重叠的多个特性)。
圣= geneSummaryTable ({“分配的”,“Unassigned_ambiguous”,“未分配的功能”}:)栏(table2array (st)”,“堆叠的”);图例(st.Properties.RowNames',“口译员”,“没有”,'地点',“东南”);包含(“样本”) ylabel (读取的数)
st = 3x4 table untreated3 untreated4 treated2 treated3 __________ __________ __________ __________ Assigned 1.5457e+07 1.6302e+07 1.5146e+07 1.8856e+07 unassignned_ambiguous 1.5708e+05 1.6882e+05 1.6194e+05 1.9977e+05 unassignned_nofeature 7.5455e+05 5.8309e+05 5.8756e+05 6.8356e+05
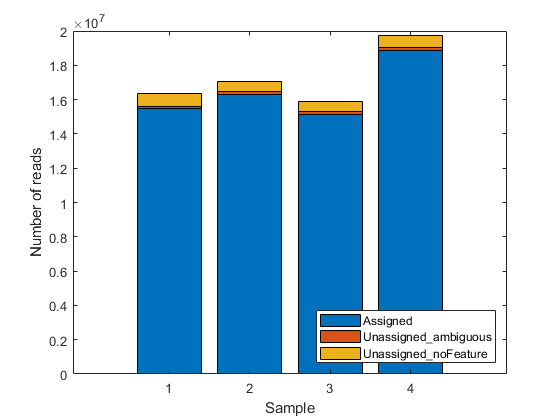
请注意,汇总表中没有报告SAM文件中的一小部分对齐记录。您可以从SAM文件中的记录总数与同一SAM文件在计数过程中处理的记录总数之间的差异中注意到这一点。这些未报告的记录对应于映射的记录参考未在GTF文件中注释的序列,因此未在计数过程中处理。如果基因模型说明了在读取映射步骤中使用的所有参考序列,则所有记录都在汇总表的一个类别中报告。
genesummarytable {“TotalEntries”,:} - 总和(genesummarytable {2:结束,:})
Ans = 89516 95885 98207 104629
绘制给定染色体的读取覆盖率
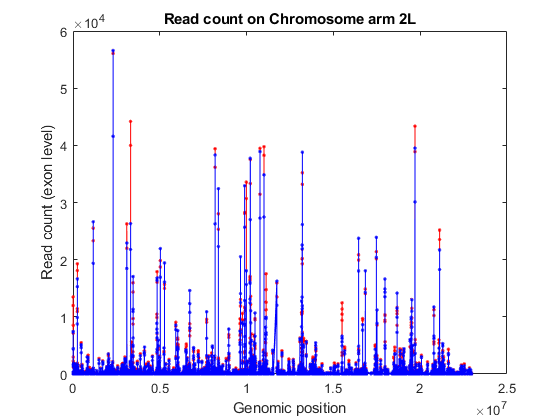
在未经使用功能的情况下执行读取计数时featurecount,默认id由属性或元特征(默认情况下为gene_id)组成,后跟特征的开始位置和停止位置(默认情况下为外显子)。您可以使用外显子开始位置绘制所考虑的任何染色体的读取覆盖率,例如染色体臂2L。
%考虑染色体臂2Lchr2L=strcmp(外计数参考,' 2 l ');exonCount = exonCountTable{: 3:结束};%检索外显子起始位置exonStart = regexp (exonCountTable {chr2L 1},“_ (\ d +) _”,“代币”);exonStart = [exonStart {}):;exonStart = cellfun(@str2num, [exonStart{:}]');按起始位置排序外显子[~,idx]=排序(exonStart);%沿着基因组坐标绘制读取覆盖率图;情节(exonStart (idx) exonCount (idx、处理),“。r”,......Exonstart(Idx),Exoncount(IDX,未经处理),“-b”);包含(“基因组的位置”);ylabel('读数(外显子水平)');头衔('读取染色体臂2L');%分别绘制每组的读取覆盖率图;次要情节(2,1,1);情节(exonStart (idx) exonCount (idx,未经处理的),“。r”);ylabel('读数(外显子水平)');头衔(染色体臂2L(处理过的样本));次要情节(2,1,2);情节(exonStart (idx) exonCount (idx、处理),“-b”);ylabel('读数(外显子水平)');包含(“基因组的位置”);头衔(“染色体臂2L(未处理样本)”);
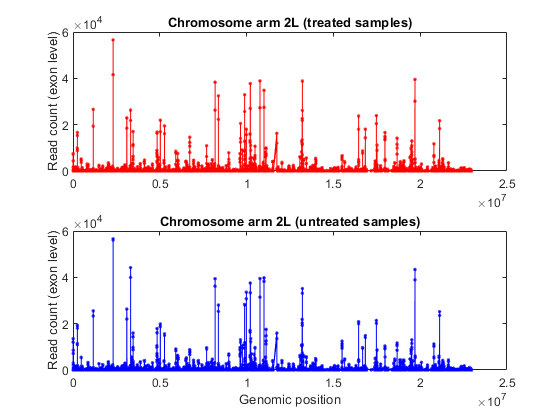
或者,考虑到给定染色体中每个基因的起始位置,可以绘制读取覆盖。文件pasilla_geneLength.mat包含一个表,其中包含相应基因注释文件中每个基因的开始和停止位置。
%负载基因启动和停止位置信息负载pasilla_geneLengthGooleHength(1:10,:)
ans = 10 x5表ID名称引用起停 _______________ ___________ _________ _____ _____ {' FBgn0037213}{‘CG12581} 3 r 380 10200{‘FBgn0000500}{“Dsk”}3 r 15388 16170{‘FBgn0053294}{‘CR33294} 3 r 17136 21871{‘FBgn0037215}{‘CG12582} 3 r 23029 30295{‘FBgn0037217}{‘CG14636} 3 r 30207 41033{‘FBgn0037218}{“辅助”}3 r 37505 53244{'FBgn0051516'} {'CG31516'} 3R 44179 45852 {'FBgn0261436'} {'DhpD'} 3R 53106 54971 {'FBgn0037220'} 3R 56475 58077 {'FBgn0015331'} {'abs'} 3R 58765 60763
%考虑染色体3(“参考”是一个分类变量)chr3 = (geneLength。参考= =' 3 l ') | (geneLength。参考= =“3 r”);总和(chr3)考虑3号染色体的基因计数counts = genecounttable {:,3:结束};[〜,j,k] =相交(GenecountTable {:,“身份证”},GeneLength {CHR3,“身份证”});gstart = geneLength {k,“开始”};gcounts =计数(j:);%根据开始位置升序排序[~, idx] = (gstart)进行排序;%按基因组位置绘制读取覆盖率图;情节(gstart (idx) gcounts (idx、处理),“。r”,......gstart (idx) gcounts (idx,未经处理的),“-b”);包含(“基因组的位置”) ylabel (“读计数”);头衔(“3号染色体上的读取计数”);
ans = 6360

正常化读数
RNA-Seq数据中的读计数已被发现与转录本[2]的丰度呈线性相关。然而,给定基因的读计数不仅取决于该基因的表达水平,还取决于测序的读计数总数和基因转录本的长度。因此,为了从读计数推断一个基因的表达水平,我们需要考虑测序深度和基因转录本长度。规范化读计数的一种常见技术是使用RPKM (read Per Kilobase Mapped)值,其中读取计数由产生的读总数(以百万为单位)和每个转录本的长度(以千碱基为单位)进行规范化。然而,这种归一化技术并不总是有效的,因为少数非常高表达的基因可以支配总通道数并使表达分析产生偏差。
更好的归一化技术包括通过考虑每个样本的尺寸因子来计算有效库大小。通过将每个样本的计数除以相应的大小因素,我们将所有计数值带到共同规模,使其具有可比性。直观地,如果样品A比样品B更深的n次,则预期非差异表达基因的读数比样品B中的样品A中的平均n倍,即使表达没有差异。
为了估计大小因素,将观察到的计数比率的中值中值取向伪参考样品的比例,其计数可以通过考虑所有样品的每个基因的几何平均值[3]来获得。然后,为了将观察到的计数转换为公共规模,将观察到的每个样本中的观察到的计数除以相应的尺寸因子。
用几何平均逐行估计伪参考pseudoRefSample = geomean(数量2);nz = pseudoRefSample > 0;比率= bsxfun (@rdivide计数(新西兰:),pseudoRefSample(新西兰));sizeFactors =值(比率,1)
SOUDEFACTORS = 0.9374 0.9725 0.9388 1.1789
%转换为普通比例normCounts=bsxfun(@rdivide,counts,sizeFactors);normCounts(1:10,:)
Ans = 1.0e+03 * 0 0.0010 0.0011 0.0017 0.1515 0.1203 0.1470 0.1120 0.0213 0.0123 0.0107 0.0161 0.0021 0.0041 0 0.0008 7.0315 5.2721 5.1225 5.1124 0.5003 0.5450 0.5241 0.4869 0.0053 0.0062 0.0107 0.0068 00 0.0021 0.0008 1.2375 1.1753 1.2122 1.2003 000 0.0008
您可以使用该功能来欣赏这种归一化的影响箱线图用来表示统计测量值,如中位数、四分位数、最小值和最大值。
图;子批次(2,1,1)MABOX图(log2(计数),“头衔”,“原始读计数”,“方向”,'水平的') ylabel (“样本”)Xlabel('日志2(计数)'次要情节(2,1,2)maboxplot (log2 (normCounts),“头衔”,'正常化读数',“方向”,'水平的') ylabel (“样本”)Xlabel('日志2(计数)')

计算平均值,色散和折叠变化
为了更好地表征数据,我们考虑标准化计数的平均值和分散。读数的方差由两个术语的总和给出:样本(原始方差)的变化和通过计数读取测量表达的不确定性(射门噪音或泊松)。对高表达基因的原始方差术语占主导地位,而射击噪声占低低调基因的噪声。您可以将经验色散值与日志比例中的归一计数的平均值绘制,如下所示。
%考虑平均值meanTreated =意味着(normCounts(:治疗),2);meanUntreated =意味着(normCounts(:,未经处理的),2);%考虑色散dispTreated=std(正常计数(:,治疗),0,2)。/meanTreated;dispUntreated=std(正常计数(:,未治疗),0,2)。/平均未治疗;%在对数尺度上绘图图;loglog(默示,饮食,“r”。);持有在;loglog(指未处理、未处理、,“b”。);包含(“log2(的意思)”);ylabel(‘log2(分散度)’); 传奇(“治疗”,“未经处理”,'地点','西南');

由于重复的次数很少,因此预期离散值会在真实值附近产生一些方差是不足为奇的。其中一些方差反映了抽样方差,另一些则反映了样本中基因表达的真实可变性。
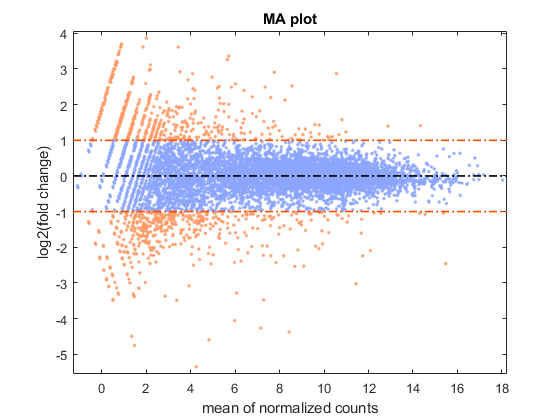
通过计算每个基因的折叠变化(Fc),即对未处理基团的计数,通过计算每个基因的折叠变化(Fc),可以查看两个条件中基因表达的差异。通常,这些比率在LOG2刻度中考虑,使得任何改变相对于零(例如,1/2或2/1的比对对应于日志比例的-1或+1)。
%计算平均值和log2FCmeanBase=(meanTreated+meanUntreated)/2;foldChange=meanTreated./meanUntreated;log2FC=log2(foldChange);%图平均值与fold change (MA图)mairplot (meanTreated meanUntreated,“类型”,“妈妈”,“Plotonly”,真);设置(获取)(gca,“包含”),'细绳',“标准化计数的平均值”)设置(get(gca,'ylabel'),'细绳',“log2(褶皱变化)”)
警告:忽略零值
可以用相应的基因名注释图中的值,交互式地选择基因,并通过调用梅尔普洛特功能如下图所示:
mairplot (meanTreated meanUntreated,“标签”, geneCountTable。ID,“类型”,“妈妈”);
警告:忽略零值
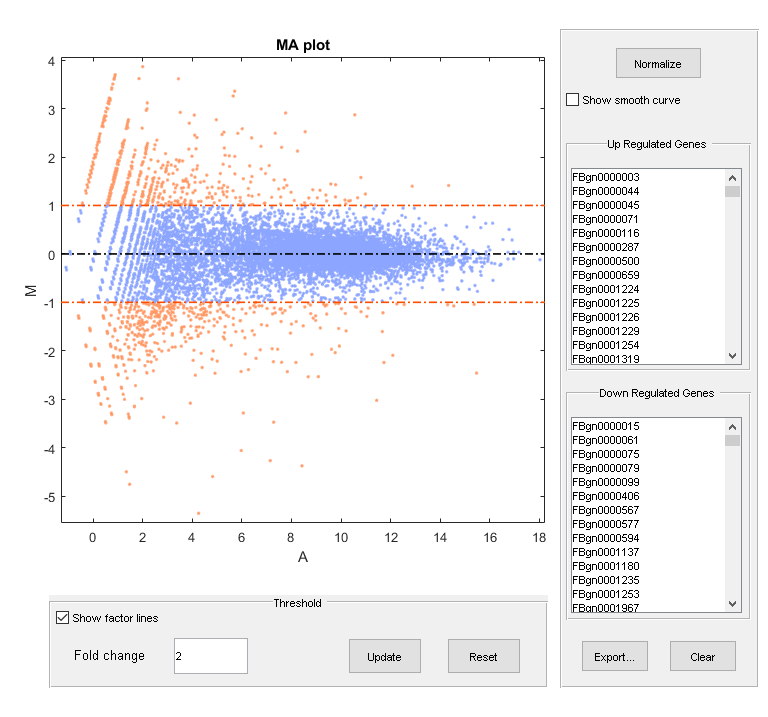
将每个基因的平均值和折数变化信息存储在表格中,方便快捷。然后,您可以通过按基因名索引表来访问关于给定基因或满足特定条件的一组基因的信息。
%创建包含每个基因统计信息的表geneTable =表(meanBase meanTreated、meanUntreated foldChange, log2FC);geneTable.Properties.RowNames = geneCountTable.ID;
% 概括总结(geneTable)
变量:平均基数:11609x1双值:最小0.21206中值201.24最大2.6789e+05平均治疗:11609x1双值:最小0中值201.54最大2.5676e+05平均未治疗:11609x1双值:最小0中值199.44最大2.7903e+05折叠更改:11609x1双值:最小0中值0.99903最大Inf log2FC:11609x1双值:最小Inf中值-0.001406最大Inf
% 预览geneTable (1:10,:)
ANS = 10x5表meanBase meanTreated meanUntreated foldChange log2FC ________ ___________ _____________ __________ ___________ FBgn0000003 0.9475 1.3808 0.51415 2.6857 1.4253 FBgn0000008 132.69 129.48 135.9 0.95277 -0.069799 FBgn0000014 15.111 13.384 16.838 0.79488 -0.33119 FBgn0000015 1.7738 0.42413 3.1234 0.13579 -2.8806 FBgn0000017 5634.6 5117.4 6151.8 0.83186 -0.26559 FBgn0000018 514.08505.48 522.67 0.96711 -0.048243 FBGN0000024 7.2354 8.7189 5.752 1.5158 0.60009 FBGN0000028 0.74465 1.4893 0 INF FBGN0000032 1206.3 1206.2 1206.4 0.99983 -0.00025093 FBGN0000036 0.21206 0.42413 0 INF INF
%获取有关特定基因的信息myGene =“FBgn0261570”;Genetable(Mygene,:) Genetable(Mygene,{“平均基数”,“log2FC”})%访问给定基因列表的信息myGeneSet = {“FBgn0261570”,“FBgn0261573”,“FBgn0261575”,“FBgn0261560”};geneTable (myGeneSet:)
ans = 1 x5表meanBase meanTreated meanUntreated foldChange log2FC ________ ___________ _____________ __________ _______ FBgn0261570 4435.5 4939.1 3931.8 1.2562 0.32907 ans = 1 x2表meanBase log2FC ________ _______ FBgn0261570 4435.5 - 0.32907 ans = 4 x5表meanBase meanTreated meanUntreated foldChange log2FC ________ ___________ _______________________ _______ FBgn0261570 4435.5 4939.1 3931.8 1.2562 0.32907 FBgn0261573 2936.9 2954.8 2919.1 1.0122 0.01753 FBgn0261575 4.3776 5.6318 3.1234 1.8031 0.85047 FBgn0261560 2041.1 1494.3 2588 0.57738 -0.7924
用负二项模型推导微分表达式
确定两种情况下的基因表达是否统计上不同,包括拒绝两个数据样本来自均值相等分布的无效假设。该分析假设读取计数根据负二项分布建模(如[3]中所述)。函数nbintest用三个可能的选项执行这种类型的假设检验,以指定方差和均值之间的联系类型。
通过指定方差与均值之间的链接,我们假设方差等于平均值,并且计数由泊松分布进行建模[4]。
tIdentity = nbintest(计数(:治疗),计数(:,未经处理的),“VarianceLink”,“身份”);H = Plotvarycelink(TIDETITY);%设置自定义标题h (1) .Title。字符串=“已处理样本的方差链接”;h (2) .Title。字符串=“未处理样本的方差链接”;

或者,通过指定方差为散粒噪声项(即均值)和常数乘以均值的平方之和,计数根据[5]中描述的分布建模。常数项使用数据中的所有行进行估计。
tConstant = nbintest(计数(:治疗),计数(:,未经处理的),“VarianceLink”,'持续的');h = plotVarianceLink (tConstant);%设置自定义标题h (1) .Title。字符串=“已处理样本的方差链接”;h (2) .Title。字符串=“未处理样本的方差链接”;


最后,将方差作为散粒噪声项(即均值)和局部回归的非参数均值平滑函数的和,根据[3]中提出的分布对计数进行建模。
tLocal = nbintest(计数(:治疗),计数(:,未经处理的),“VarianceLink”,“LocalRegression”);h = plotVarianceLink (tLocal);%设置自定义标题h (1) .Title。字符串=“已处理样本的方差链接”;h (2) .Title。字符串=“未处理样本的方差链接”;


为了评估哪种拟合最适合所考虑的数据,可以比较单个图中的拟合曲线,如下所示。
h=绘图变量链接(T局部,“比较”,真的);%设置自定义标题h (1) .Title。字符串=“已处理样本的方差链接”;h (2) .Title。字符串=“未处理样本的方差链接”;


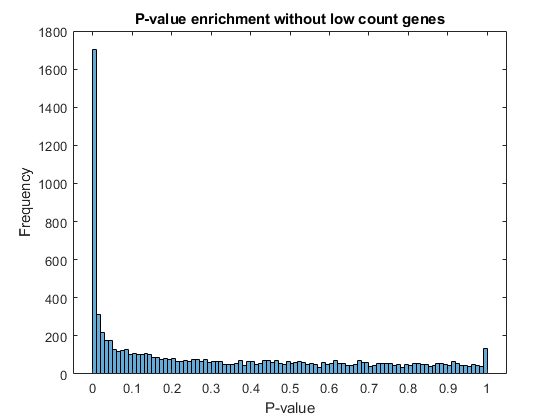
产量nbintest包括一个P值向量。P值表示在无效假设下出现与观察到的表达一样强烈(甚至更强)的表达变化的概率,即条件对基因表达没有影响。在P值直方图中,我们观察到低值的富集(由于差异表达基因),而其他值均匀分布(由于非差异表达基因)。值等于1的富集是由于计数非常低的基因。
图;直方图(tLocal.pValue 100)包含('p值') ylabel (“频率”)标题(“假定值浓缩”)

过滤掉那些计数相对较低的基因,以观察非显著性P值在整个范围(0,1)内更均匀的分布。注意,这不会影响显著性P值的分布。
lowCountThreshold = 10;lowCountGenes = all(count < lowCountThreshold, 2);直方图(tLocal.pValue (~ lowCountGenes), 100)包含('p值') ylabel (“频率”)标题(“无低计数基因的p值富集”)

多个测试和调整的P值
由于多重测试问题,对p值设置阈值以确定哪些倍数的更改比其他更改更重要,对于这种类型的数据分析是不合适的。当执行大量同时进行的测试时,仅仅由于机会而获得重要结果的概率会随着测试数量的增加而增加。为了考虑多个测试,对p值进行修正(或调整),使由于偶然而观察到至少一个显著结果的概率仍然低于期望的显著性水平。
Benjamini-Hochberg(BH)调整[6]是一种统计方法,它提供了一个调整后的P值,回答了以下问题:如果所有调整后的P值低于给定阈值的基因都被认为是显著的,那么假阳性的比例是多少?设置调整后的p值的阈值0.1,相当于认为10%个假阳性为可接受的,并且通过考虑所有低于调节阈值的基因的p值来识别被显著表达的基因。
%计算调整后的p值(BH校正)padj = mafdr (tLocal.pValue,“BHFDR”,真的);%添加到现有表中geneTable.pvalue=tLocal.pvalue;geneTable.padj=padj;%创建一个包含重要基因的表sig = geneTable。padj < 0.1;: geneTableSig = geneTable(团体);geneTableSig = sortrows (geneTableSig,'padj');numberSigGenes=大小(geneTableSig,1)
numberSigGenes = 1904
识别最上调和下调的基因
现在,你可以通过考虑在选定的截止点之上的绝对折叠变化来确定最上调或下调的基因。例如,在log2规模中截断1,就会产生具有2倍变化的上调基因列表。
%发现上调基因Up = genetablesig.log2fc> 1;Upgenes = Sortrows(Genetablesig(UP,:),“log2FC”,“下”);numberSigGenesUp = sum ()%显示前10位上调基因: top10GenesUp = upGenes (1:10)%寻找下调基因向下= genetablesig.log2fc <-1;DownGenes = Sortrows(Genetablesig(Down,:),“log2FC”,“提升”); numberSigGenesDown=总和(向下)%找到前10个下调基因top10genesdown = downgenes(1:10,:)
numberSigGenesUp = 129 top10GenesUp = 10x7 table meanBase meanTreated mean未经处理foldChange log2FC pvalue padj ________ ___________ _____________ __________ ______ __________ __________ FBgn0030173 3.3979 6.7957 0 Inf Inf 0.0063115 0.047764 FBgn0036822 3.1364 6.2729 0 Inf Inf 0.012203 0.079274 FBgn0052548 8.158 15.269 1.0476 14.575 3.86540.00016945 0.0022662 FBgn0050495 6.8315 12.635 1.0283 12.287 3.6191 0.0018949 0.017972 FBgn0063667 20.573 38.042 3.1042 12.255 3.6153 8.5037e-08 2.3845e-06 FBgn0033764 91.969 167.61 16.324 10.2680.024283 FBgn0037191 7.1766 12.753 1.6003 7.9694 2.9945 0.0047803 0.038193 FBgn0033943 6.95 12.319 1.581 7.7921 2.962 0.0053635 0.041986 numberSigGenesDown = 181 top10GenesDown = 10x7 table meanBase meanTreated mean未经处理foldChange log2FC pvalue padj ________ ___________ _____________ __________ _______ __________ __________ FBgn0053498 15.46930.938 0 0负9.8404 e-11 4.345 e-09 FBgn0259236 0.00027393 6.8092 13.618 0负1.5526 e-05 FBgn0052500 4.3703 0 8.7405 7.3908 0负0.00066783 - 0.0075343 FBgn0039331 3.6954 0 0负0.0019558 - 0.018474 FBgn0040697 3.419 0 6.8381 5.8291 0负0.0027378 - 0.024336 FBgn0034972 2.9145 0 0负0.0068564 - 0.05073 FBgn0040967 2.6382 0 5.2764 0负无穷0.0096039 0.065972 FBgn0031923 2.3715 0 4.7429 0 -Inf 0.016164 0.098762 0.024421 2.9786 121.97 0.024421 -5.3557 5.5813e-33 1.5068e-30
通过以对数标度绘制折叠变化与平均值的关系,并根据调整后的P值对数据点进行着色,可以很好地可视化基因表达及其意义。
图2散点图(log2(geneTable.meanBase)、geneTable.log2FC、3、geneTable.padj、,“哦”) colormap (flipud(酷(256)))colorbar;ylabel('log2(折叠变化)')Xlabel('log2(标准化计数的平均值)')标题('折叠通过fdr'改变')
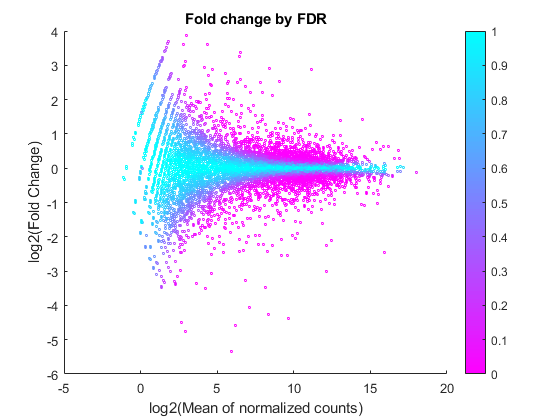
你可以在这里看到,对于弱表达基因(即那些平均值低的基因),FDR通常是高的,因为低读计数由泊松噪声主导,因此,任何生物变异都淹没在读计数的不确定性中。
工具书类
[1] Brooks等人。果蝇和哺乳动物之间的RNA调节贴图的保护。Genome Research 2011. 21:193-202。
Mortazavi等。通过RNA-Seq定位和量化哺乳动物转录组。2008年自然方法。5:621 - 628。
[3] Anders等人。序列计数数据的差异表达分析。基因组生物学2010. 11:R106。
[4] Marioni等人。RNA-Seq:技术再现性评估和与基因表达阵列的比较。基因组研究2008。18:1509-1517。
[5] Robinson等人。评估标记丰度差异的缓和统计检验。生物信息学2007。23(21):2881-2887。
[6] Benjamini等人,《控制错误发现率:一种实用而有效的多重测试方法》,1995年,《皇家统计学会杂志》,B57(1):289-300系列。
另见
featurecount|梅尔普洛特|nbintest|plotVarianceLink

