从roi和蒙版创建带标签的阻塞图像
这个例子展示了如何从一组roi创建一个标记块图像。
在本例中,您使用两种方法来获取和显示标记数据。一种方法是使用多边形ROI对象存储肿瘤和正常组织区域边界的坐标。的polyToBlockedImage函数将ROI坐标转换为标记的块图像。第二种方法是使用掩模将图像分割为组织和背景。这个例子结合了ROI和蒙版图像中的信息,创建了一个带有数字像素标签的单一块图像,对应于肿瘤、正常组织和背景区域。
使用CAMELYON16数据集中包含肿瘤组织的淋巴结训练图像的修改版本创建块图像。修改后的图像有三个粗分辨率。空间参考已经被调整,以加强一致的宽高比,并在每个级别注册特征。
bim = blockedImage (“tumor_091R.tif”);
标签数据加载
CAMELYON16数据集提供了肿瘤和正常区域的标签,作为一组坐标,指定了相对于最佳分辨率级别的手工标注区域边界。当像素同时存在于正常区域和肿瘤区域的边界内时,这些像素的正确标记就是正常组织。
为阻塞的图像加载标签数据。的标签的修改版本tumor_091.tifCAMELYON16数据集的图像。原始标签以XML格式存储。修改后的标签已被重新采样并保存为MAT文件。
roiPoints =负载(“labelledROIs.mat”)
roiPoints =结构体字段:nonCancerRegions: {[46×2 double]} cancerRegions: {6×1 cell}
将标签表示为ROI对象
创建多边形ROI对象,存储肿瘤边界和正常组织边界的坐标。
tumorpolyys = cellfun(@(position) images.roi.Polygon(...位置=位置,可见=“上”颜色=“r”),...roiPoints.cancerRegions);normalpolyys = cellfun(@(position) images.roi.Polygon(...位置=位置,可见=“上”颜色=“g”),...roiPoints.nonCancerRegions);
在原始数据上显示roi和标签
显示覆盖多边形roi的图像。由于roi与图像具有相同的坐标系统,因此改变显示图像的分辨率仍然能够准确地呈现roi。
h = bigimageshow (bim);集(tumorPolys、家长= gca);集(normalPolys、家长= gca);标题(“分辨率水平:“+ num2str (h.ResolutionLevel));

放大一个ROI。肿瘤区域边界为红色,周围为正常组织内部的绿色区域。
ylim([26803010]) title(“分辨率水平:“+ num2str (h.ResolutionLevel));

创建标记的ROI数据块图像
使用polyToBlockedImage函数创建ROI坐标数据的标记块图像。的polyToBlockedImage函数需要ROI坐标、ROI标签和输出块图像的大小作为输入。
获得xy -协调正常区域和肿瘤区域的数据,并将它们合并成一个单一的数据roiPositions单元阵列。
normalRegions = roiPoints.nonCancerRegions;tumorRegions = roiPoints.cancerRegions;roiPositions = [normalRegions;tumorRegions];
找出正常区域和肿瘤区域的数量,并分配标签1到正常组织和标签2肿瘤组织。按指定的顺序分配标签roiPositions.标签指定为uint8值以减少存储所需的内存。
numNormalRegions =元素个数(normalRegions);numTumorRegions =元素个数(tumorRegions);roiLabelIDs = [(numNormalRegions 1“uint8”);2 * 1 (numTumorRegions 1“uint8”));
为新阻塞的图像选择所需的分辨率级别。这个选择是效率和准确性之间的权衡。使用较粗的分辨率级别可减少处理时间和存储大小。使用更精细的分辨率级别可以增加掩码中保留的细节级别。粗分辨率级别可以用于规则的roi,如多边形。对于小的徒手roi,精细的分辨率级别可能更合适。对于本例,使用中间分辨率级别。
maskLevel = 2;
指定新阻塞图像的图像大小以匹配原始图像的图像大小,荡妇,在所需的分辨率级别。
图象尺寸= bim.Size (maskLevel 1:2);
创建一个有标签的阻塞图像。保持原始块图像的空间引用,荡妇,在所需的分辨率级别。默认情况下,不属于任何ROI的像素被分配为数字标签0.
图象尺寸,bROILabels = polyToBlockedImage (roiPositions roiLabelIDs BlockSize = bim.BlockSize (maskLevel 1:2),...WorldStart = bim.WorldStart (maskLevel 1:2), WorldEnd = bim.WorldEnd (maskLevel 1:2));
ROI标签和原始数据的叠加显示
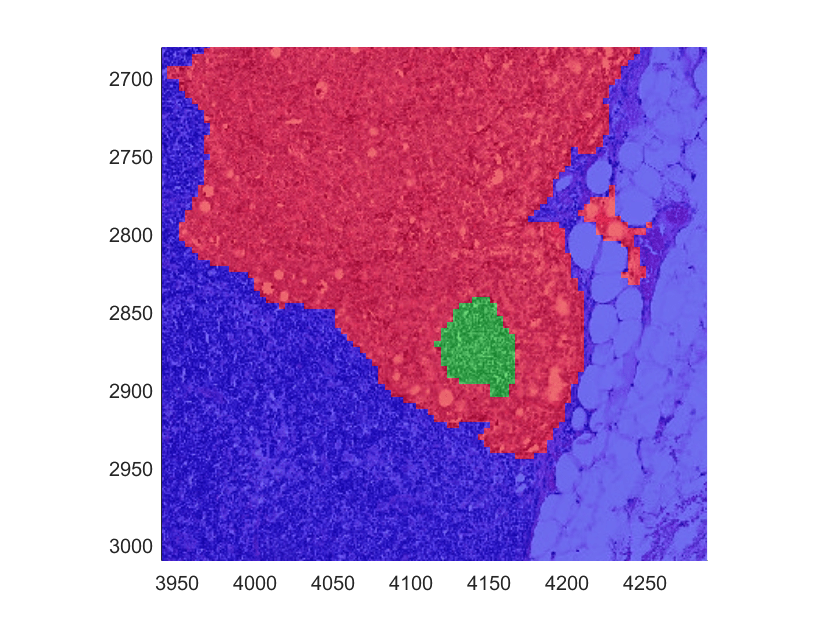
显示覆盖在原始图像上的标记阻塞图像。红色显示肿瘤区域,绿色显示完全被肿瘤包围的正常组织区域。背景及与背景相连的正常组织以蓝色显示,说明连接的正常区域被错误地归类为背景。
hbim = bigimageshow (bim);showlabels (hbim bROILabels Colormap = [0 0 1;0 1 0;1 0 0]);

放大显示为绿色的正常组织区域。可视化地验证ROI边界是否有足够的细节表示。
ylim([2680 3010])
使用蒙版分离组织和背景
使用图像分割创建一个正确区分正常组织和背景的遮罩。由于阈值要求将底层图像数据读入内存,背景和前景区域足够大,所以最粗的分辨率是合适的。面具是1(真正的),用于灰度值小于130的像素。填充小洞的掩模执行形态关闭使用bwmorph函数。
btissueMask =应用(荡妇,...@ (bs) bwmorph (rgb2gray (bs.Data) < 130“关闭”),级别= 3);bigimagehow(btissuemask);

创建单个标记阻塞图像
将组织掩模和ROI标记数据组合成最终标记的阻塞图像。尽管已经在比组织掩模更精细的分辨率水平创建了ROI图像,但是它们可以处理它们,因为它们源自相同的图像并具有相同的世界范围。使用combineLabelsHelper函数,在本例的最后定义。
bLabels =应用(bROILabels、@combineLabels ExtraImages = btissueMask);
显示覆盖在原始图像上的标记阻塞图像。三个标签(正常、肿瘤和背景)分别以绿色、红色和蓝色显示。
hbim = bigimageshow (bim);showlabels (hbim bLabels Colormap = [0 0 1;0 1 0;1 0 0]);标题(“背景(蓝色),正常组织(绿色),肿瘤(红色)”)

放大一个感兴趣的区域,以更详细地检查。
ylim([2680 3010])

金宝app支持函数
combineLabels将基于roi的标签图像和基于掩码的标签图像合并为单个标签图像。
函数btissueMask blabel = combineLabels (bs)%纸巾遮罩块尺寸较小btissueMask = imresize (btissueMask、大小(bs.Data));%转换为标签,0是背景,1是前景。blabel = uint8 (btissueMask);%用2标记肿瘤区域blabel (bs.Data = = 2) = 2;结束
另请参阅
blockedImage|bigimageshow|blockedImageDatastore

