主要内容
sbiosmulate.
模拟SimBiology模型
语法
描述
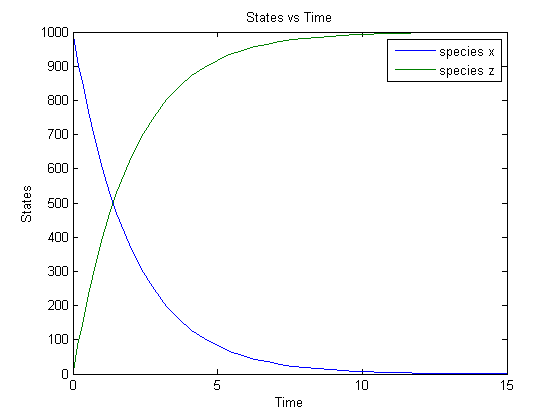
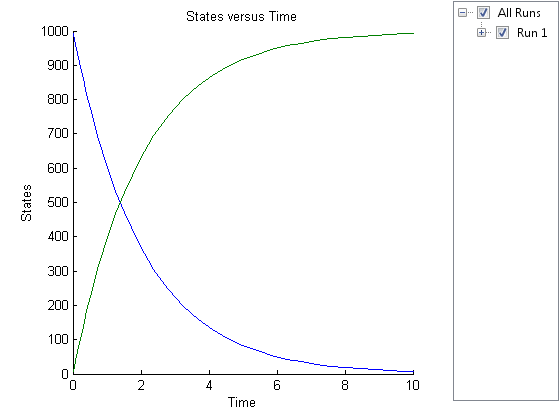
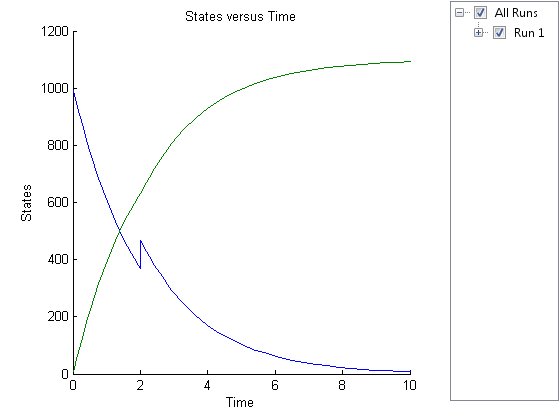
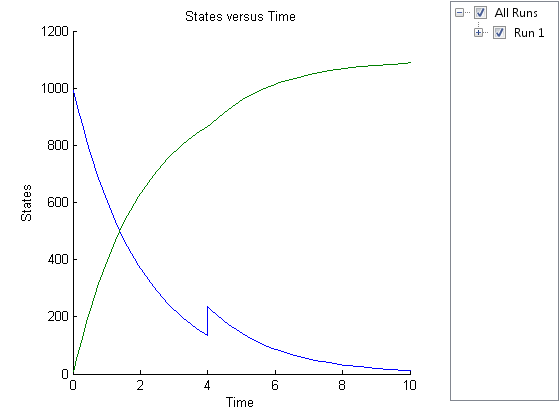
simdataobj.= sbiosimulate (___)辛迪塔对象simdataobj.使用前面的语法中的任何输入参数。
例子
输入参数
输出参数
另请参阅
addconfigset|Configset对象|getconfiget.|模型对象|重复糖对物体|Sbioaccelerate.|sbiomodel|scheduldose对象|setAceConfigset.|辛迪塔对象|变体对象
在R2006A之前介绍