createSimFunction(模型)
创建SimFunction对象
语法
描述
F= createImfunction(模型,参数个数,可见,服用)SIMFUNCTIP对象F你可以像函数句柄一样执行。的参数个数和可见参数定义函数的输入和输出F当它被执行时,并且服用定义物种的剂量信息。看到SIMFUNCTIP对象有关如何执行的详细信息F.
请注意
忽略模型的活性剂量和变体时
F执行。F在创建后是不可变的。F除非您设置,否则在第一个函数执行时自动加速'autoaccelerate'到假.如果希望在部署应用程序中加速对象,则手动加速该对象。
输入参数
模型- - - - - -SimBiology模型
Simbiology模型对象
SimBiology模型,指定为SimBiology模型对象.
函数使用相同的configset.设置通过复制configset.对象的目的模型对象。然而,该函数忽略以下内容configset.设置:Statestolog.,产出,StopTime, 和SensitivityAnalysisOptions因为这些设置是由函数的其他输入提供的。
参数个数- - - - - -SIMF功能的输入F
字符向量|字符向量的单元格阵列|{}|SimBiology。场景对象
SIMF功能的输入F,指定为字符向量,字符向量单元数组,空单元数组{}, 要么SimBiology。场景对象。字符向量表示定义输入的模型数量(物种、隔间或参数)的名称F.使用空单元格数组{}或空场景对象simbiology.scenarios.prevey()创建一个SIMFUNCTIP对象没有参数。
要明确地命名模型数量,请使用限定名称,其中包括单元的名称。若要命名反应作用域的参数,请使用反应名称来限定参数。如果名称不是有效的MATLAB®变量名称,围绕方括号括起来[反应1]。[参数1].
服用- - - - - -剂量的种类或剂量物体
字符向量|字符向量的单元格阵列|剂量客体矢量|[]
剂量种类或剂量对象,指定为字符向量、字符向量单元阵列、剂量对象向量或空数组[].
如果它是[],没有物种被剂量在模拟期间,除非你指定场景具有在其条目中定义的剂量的对象。
如果它是字符向量的小区数组,则必须是1-by-N阵列,其中N是给药的次数。如果打算使用,您可以使用重复的物种名称多剂量当您运行SIMF功能时,对于相同的物种F.仅使用时,使用物种名称不包含有关剂量特性的信息。如果您有一个包含的剂量对象参数化属性如量,使用剂量对象作为输入,而不仅仅是物种名称,以便将此类参数信息传递给创建的SimFunctionF.
如果它是剂量对象的向量,它必须是1-by-N向量,N为剂量对象的数量。如果剂量对象具有非默认数值的属性,则忽略这些值并发出警告。只有TargetName,DurationParameterName,LagParameterName, 和参数化属性用于创建SimFunction对象F,也就是说,定义给的属性F.详细了解如何给属性表被填充,查看财产摘要.
的创建期间指定的剂量信息simfunction.对象必须与您在执行对象的执行期间指定的给药信息一致。换句话说,元素的数量给的属性simfunction.F必须等于输入的剂量之和场景对象φ和剂量输入参数u当你执行对象时。
变体- - - - - -替代模型值
变体对象|变体对象矢量
替代模型值,指定为变量对象的变量或向量。这些值应用于模型基线值时simfunction.创建对象。如果有多个变体引用相同的模型元素,则使用最后一次发生。
名称-值对的观点
指定可选的逗号分隔的对名称,值参数。名称参数名和价值是相应的价值。名称必须出现在引号内。可以以任意顺序指定多个名称和值对参数Name1, Value1,…,的家.
'使用反应',真实指定执行SimFunctionF在平行下。
“UseParallel”- - - - - -旗帜执行SIMFUNCTIONF并行
假(默认)|真的
旗帜执行SIMFUNCTIONF并行,指定为逗号分隔的配对组成“UseParallel”和真的要么假.如果真的和并行计算工具箱™可用,SimFunctionF并行执行。
例子:“UseParallel”,真的
'autoaccelerate'- - - - - -旗帜加速第一次评估模型simfunction.
真的(默认)|假
旗帜加速模型的第一次评估simfunction.对象,指定为逗号分隔的对,由'autoaccelerate'和真的要么假.
将值设置为假如果你有一个模型,因为模型的加速可能会比模型的实际模拟花费更长的时间,所以要快速模拟。
例子:'autoaccelerate',假
“SensitivityOutputs”- - - - - -灵敏度输出因素
{}(默认)|字符向量的单元格阵列|“所有”
灵敏度输出因子,指定为逗号分隔对组成“SensitivityOutputs”以及字符向量的单元格数组。字符向量是需要计算灵敏度的模型数量(物种和参数)的名称。默认值是{}这意味着没有输出因素。输出因子是时间相关导数的分子偶像生物学的敏感性分析.
使用关键字“所有”要么“所有”将所有型号的数量指定为灵敏度输出。然而,{'全部'}表示名为的型号数量全部在模型中。[“全部”,“x”]为命名的物种设置灵敏度输入因子或输出因子全部和x.
您必须指定两者“SensitivityOutputs”和“SensitivityInputs”用于敏感性计算的名称值对参数。
例子:“SensitivityOutputs”,“所有”
“SensitivityInputs”- - - - - -输入因素的敏感性
{}(默认)|字符向量的单元格阵列|“所有”
灵敏度输入因子,指定为逗号分隔对组成“SensitivityInputs”以及字符向量的单元格数组。角色向量是要计算灵敏度的模型数量(物种、隔间和参数)的名称。默认值是{}这意味着没有输入因素。输入因子是时间相关的导数的分母偶像生物学的敏感性分析.
使用关键字“所有”要么“所有”将所有型号的数量指定为灵敏度输出。然而,{'全部'}表示名为的型号数量全部在模型中。[“全部”,“x”]将灵敏度输入或输出设置为所命名的物种全部和x.
您必须指定两者“SensitivityOutputs”和“SensitivityInputs”用于敏感性计算的名称值对参数。
例子:“SensitivityInputs”,{'反应1.C1','反应1.C2'}
“SensitivityNormalization”- - - - - -计算灵敏度的归一化
'没有任何'(默认)|'一半|“全部”
计算灵敏度的归一化,指定为逗号分隔对,由“SensitivityNormalization”和'没有任何',“一半”, 要么“全部”.
'没有任何'—不规格化(默认)“一半”- 仅相对于分子的标准化“全部”- 完全献血化
有关详细信息,请参见归一化.
例子:“SensitivityNormalization”,“全部”
输出参数
F——SimFunction
simfunction.对象|SimFunctionSensitivity对象
simfunction,作为一个返回SIMFUNCTIP对象要么SimFunctionSensitivity对象.你可以执行F比如函数句柄。
F是A.SimFunctionSensitivity对象如果指定非空“SensitivityOutputs”和“SensitivityInputs”名称-值对参数。
例子
创建一个simfunction对象
这个例子使用了一个具有一级反应的放射性衰变模型
, 在哪里x和z是物种c是正速率常数。
加载包含放射性衰减模型的示例项目m1.
sbioloadproject.radiodecay;
创建一个SIMFUNCTIP对象,指定参数Reaction1.c和物种x作为没有给药物种的功能的输出。
f = createSimFunction (m1,'反应1.C',“x”[])
f = SimFunction参数:值类型单位名称 _______________ _____ _____________ ____________ {' Reaction1.c“0.5}{“参数”}{' 1 /二}可见:单位名称类型 _____ ___________ ____________ {' x '}{“物种”}{}“分子”前:没有
如果UnitConversion.选项设置为假当。。。的时候simfunction.对象f创建时,该表不显示模型数量的单位。
为了说明这一点,首先设置UnitConversion.选择假.
c = getconfigset (m1);cs.CompileOptions.UnitConversion = false;
创建simfunction.对象如之前,并注意变量命名单位就消失了。
f = createSimFunction(m1, {)'反应1.C'},{“x”},[])
f = SimFunction参数:名称值类型_______________ _____ ______________ {'ression1.c'}观察到:名称类型_____ ___________ {'x'} {'steies'}给人:没有
如果模型中的任何物种正在被剂量化,则将剂量化物种的名称指定为最后一个参数。例如,如果物种x,指定它为最后一个参数。
f = createSimFunction(m1, {)'反应1.C'},{“x”},“x”)
f = SimFunction参数:名称值类型_______________ _____ ______________ {'ression1.c'}观察到
一旦simfunction.对象创建时,可以像函数句柄一样执行它,并执行参数扫描(如果并行计算工具箱™可用,则并行执行)、蒙特卡罗模拟,并使用多个或向量化剂量进行扫描。看到SIMFUNCTIP对象有关详细信息。
使用带量信息创建一个SIMFUNCTING对象
此示例创建一个simfunction.对象具有使用剂量的给药信息RepeatDose要么ScheduleDose这些对象的对象或向量。但是,如果任何剂量对象包含如此数据开始时间,量, 和率,则忽略此类数据,并发出警告。只使用可用的数据TargetName,LagParameterName, 和DurationParameterName剂量物体的。
加载包含放射性衰减模型的示例项目m1.
sbioloadproject.radiodecay;
创建一个RepeatDose对象并指定其属性。
rdose = sbiodose ('rd');rdose.targetName =.“x”;rdose.starttime = 5;rdose.timeUnits =.“第二”;rdose.amount = 300;rdose.amountunits =.“分子”;rdose.rate = 1;rdose.RateUnits =.“分子/秒”;rdose。间隔= 100;rdose。RepeatCount = 2;
向模型中添加延迟参数和持续时间参数。
lagPara = addparameter (m1,'lp');lagPara。价值=1;lagPara。价值单位=“第二”;duropara = addparameter(m1,“迪拜”);duraPara。价值=1;duraPara。价值单位=“第二”;
将这些参数设置为剂量对象。
rdose。LagParameterName ='lp';rdose。DurationParameterName =“迪拜”;
创建一个simfunction.对象f使用RepeatDose对象rdose.你刚刚创造的。
f = createSimFunction (m1, {'反应1.C'},{“x”,“z”},rdose)
警告:剂量中的一些剂量对象具有数据。此数据将被忽略。>在SIMFunction> SIMFunction.Simfunction中的SimFunction> SIMFunction。374 F = SimFunction参数:名称值类型单位___________ ______________ __ xpression1 /秒'观察名称______________________________ 'x' 'species' 'molecule' 'z' 'species' 'molecule' Dosed: TargetName TargetDimension __________ _______________________________ 'x' 'Amount(e.g. mole or molecule)' DurationParameterName DurationParameterValue _____________________ ______________________ 'dp' 1 DurationParameterUnits LagParameterName ______________________ ________________ 'second' 'lp' LagParameterValue LagParameterUnits _________________ _________________ 1 'second'
出现警告消息,因为rdose.对象包含数据(开始时间,量,率)被忽略createSimfunction.方法。
Lotka-Volterra模型扫描参数
的不同签名SIMFUNCTIP对象模拟和扫描Gillespie描述的Lotka-Volterra(Predator-Prey)模型的参数[1]。
加载包含模型的示例项目m1.
sbioloadproject.Lotka.;
创建SimFunction对象f与c1和c2作为要扫描的输入参数,和y1.和Y2.作为没有给药物种的功能的输出。
f = createSimFunction (m1, {'反应1.C1','反应2.c2'},{'y1','y2'},[])
f = simfunction参数:名称值类型________________ _____ ___t__________ {'ression1'} {'ression'ul'} {'ression2.c2'} 0.01 {'参数'}观察到:名称类型______ ___________ {'y1'} {'物种'} {'y2'} {'speies'}给人:没有
定义一个输入矩阵,其中包含每个参数的值(c1和c2)每个模拟。行数表示仿真总数,每个模拟使用每行中指定的参数值。
Phi = [10 0.01;十0.02);
运行模拟直到停止时间为5并绘制模拟结果。
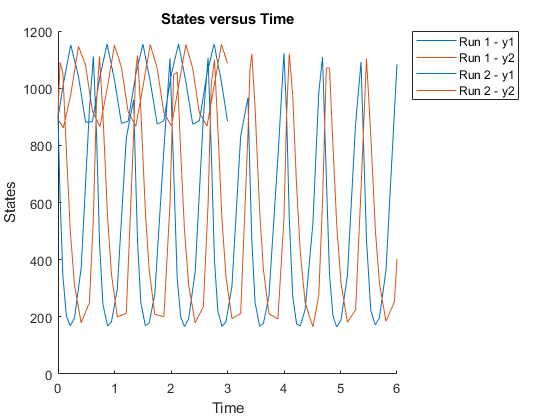
sbioplot (f(φ,5));

您还可以为每个模拟指定一个表示不同停止时间的向量。
t_stop =(3、6);sbioplot (f(φ,t_stop));
接下来,将输出时间指定为向量。
t_output = 0:0.1:5;sbioplot (f(φ,[][],t_output));
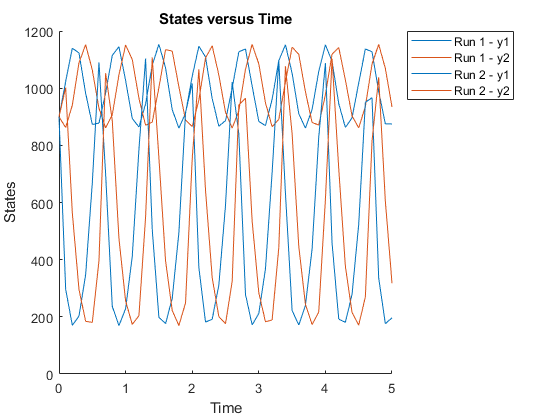
将输出时间指定为传感器的单元格数组。
T_output = {0:0.01:3, 0:0.2:6};Sbioplot (f(phi, [], [], t_output));

计算敏感性使用SimFunctionSensitivity对象
这个例子展示了如何计算Lotka-Volterra模型中某些物种的敏感性SimFunctionSensitivity对象。
加载示例项目。
sbioloadproject.Lotka.;
定义输入参数。
params = {'反应1.C1','反应2.c2'};
定义观察到的物种,它们是模拟的输出。
可见= {'y1','y2'};
创建一个SimFunctionSensitivity对象。将敏感性输出因子设置为所有物种(y1.和Y2.)在中指定可见论证和输入因素的那些参数个数参数(c1和c2),将名称-值对参数设置为“所有”.
f = createSimFunction (m1, params,可见,[],“SensitivityOutputs”,“所有”,“SensitivityInputs”,“所有”,“SensitivityNormalization”,“全部”)
f = simfunction参数:名称值类型________________ _____ ___t__________ {'ression1'} {'ression'ul'} {'ression2.c2'} 0.01 {'参数'}观察到:名称类型______ ___________ {'y1'} {'物种'} {'y2'} {'speies'}给人:没有Sensitivity Input Factors: Name Type ________________ _____________ {'Reaction1.c1'} {'parameter'} {'Reaction2.c2'} {'parameter'} Sensitivity Output Factors: Name Type ______ ___________ {'y1'} {'species'} {'y2'} {'species'} Sensitivity Normalization: Full
通过执行对象来计算敏感性c1和c2分别设置为10和0.1。将输出次数从1设置为10。t包含时间点,y包含仿真数据,并且sensMatrix灵敏度矩阵是否包含的灵敏度y1.和Y2.关于c1和c2.
[t,y,sensmatrix] = f([10,0.1],[],[],1:10);
检索时间点5的灵敏度信息。
temp = sensmatrix {:};sensmatrix2 = temp(t {:} == 5,::);sensmatrix2 =挤压(SENSMATRIX2)
sensMatrix2 =2×237.6987 -6.8447 -40.2791 5.8225
行的行sensMatrix2表示输出因子(y1.和Y2.).列表示输入因子(c1和c2).
将停止时间设置为15,无需指定输出时间。在这种情况下,输出时间是默认情况下的求解时间点。
SD = F([10,0.1],15);
从中检索计算的敏感性辛迪塔对象SD..
[t、y、输出、输入]= getsensmatrix (sd);
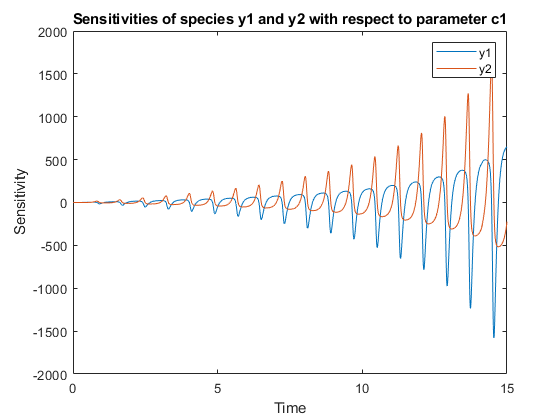
绘制物种的敏感性图y1.和Y2.关于c1.
图;情节(t y (:,: 1));传奇(输出);标题('物种Y1和Y2的敏感性相对于参数C1');包含(“时间”);ylabel('灵敏度');
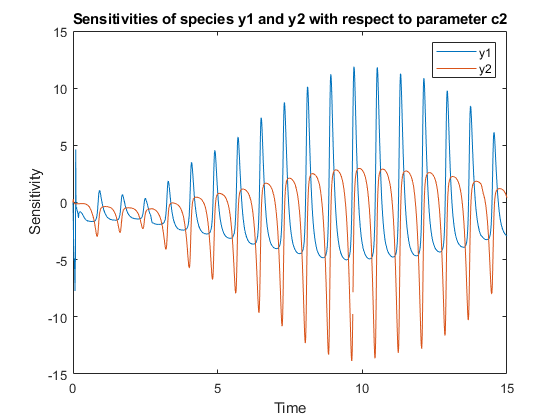
绘制物种的敏感性图y1.和Y2.关于c2.
图;情节(t y (:,: 2));传奇(输出);标题(“物种Y1和Y2关于参数C2的敏感性”);包含(“时间”);ylabel('灵敏度');
或者,您可以使用sbioplot..
SBIOPLOT(SD);
![图中包含一个坐标轴。标题为“状态与时间”的轴包含6个类型为line的对象。这些对象表示y1, y2, d[y1]/d[Reaction1]。c1), d (y2) / d (Reaction1。c1), d (y1) / d (Reaction2。c2],d[y2]/d[Reaction2.c2].](http://www.tatmou.com/help/examples/simbio/win64/CalculateSensitivitiesUsingSimFunctionSensitivityObjectExample_03.png)
您还可以使用计算的敏感性的时间积分绘制灵敏度矩阵y1.和Y2..绘图表示y1.和Y2.对c1比c2.
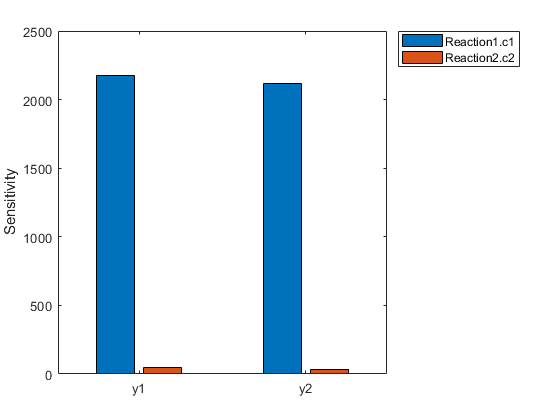
[〜,进出] =尺寸(y);结果=零(进出);为i = 1:为j = 1:out结果(i,j)= trapz(t(:),abs(y(:,i,j)));结束结束图;hbar = bar(结果);haxes = hbar(1).parent;haxes.xtick = 1:长度(输出);haxes.xticklabel =输出;传奇(投入,'地点',“NorthEastOutside”);ylabel('灵敏度');
模拟不同初始条件的葡萄糖 - 胰岛素反应模型
该实施例显示了如何模拟正常和糖尿病受试者的葡萄糖 - 胰岛素反应。
加载葡萄糖胰岛素反应模型。有关模型的详细信息,请参阅背景部分模拟葡萄糖 - 胰岛素反应.
sbioloadproject (“insulindemo”,'m1')
该模型包含不同的初始条件,存储在不同的变体中。
变量= getvariant (m1);
了解2型糖尿病患者的初始情况。
类型2 =变体(1)
Type2 = Simbiology Variant - 2型糖尿病(无效)ContentIndex:类型:名称:属性:值:1参数等离子体体积...值1.49 2参数K1值0.042 3参数K2值0.071 4参数等离子体体积...值0.04 5参数M1值0.379 6参数M2值0.673 7参数M4值0.269 8参数M5值0.0526 9参数M6值0.8118 10参数Hepatic Extrac ...值0.6 11参数kmax值0.0465 12参数kmin值0.0076 13 1 13参数kabs值0.023 14参数KGRI值0.0465 15参数F值0.9 16参数值6E-05 17参数B值0.68 18参数C值0.00023 19参数D值0.09 20参数胃glu ...值125 21参数KP1值3.09 22参数KP2值0.000723参数KP3值0.005 24参数KP4值0.0786 25参数ki值0.0066 26参数[IND IND GLU U ...值1 27参数VM0值4.65参数VMX值0.034 29参数km值466.21 30参数P2U VA参数P2U VA参数P2U VALue 0.084 31参数k值0.99 32参数alpha值0.013 33参数β值0.05 34参数伽玛值0.5 35参数ke1值0.0007 36参数Ke2值269 37参数基础等离子体g ...值164.18 38参数基础等离子体I ...价值54.81.
禁止模拟期间发出的信息性警告。
warnSettings =警告(“关闭”,“SimBiology: DimAnalysisNotDone_MatlabFcn_Dimensionless”);
创建SIMFUNCTION对象以模拟正常和糖尿病受试者的葡萄糖胰岛素响应。
指定空数组
{}对于第二个输入参数,表示将使用基本参数值模拟模型(即不执行参数扫描)。指定血糖和胰岛素浓度作为响应(要绘制的函数的输出)。
指定物种
剂量作为剂量的物种。这种物质代表模拟开始时葡萄糖的初始浓度。
normSim = createSimFunction (m1, {},......{“(等离子Glu浓缩)”,“(血浆Ins浓缩的)”},'剂量')
normSim = SimFunction参数:可见:名字类型的单位 _____________________ ___________ _______________________ {'[ 等离子体Glu浓缩的]}{“物种”}{毫克/分升的}{'[血浆Ins浓缩的]}{“物种”}{“皮摩尔/升”}给:TargetName TargetDimension __________ _____________________ {' 剂量的}{的质量(例如,克)}
对于糖尿病患者,使用变体指定初始条件Type2..
diabSim = createSimFunction (m1, {},......{“(等离子Glu浓缩)”,“(血浆Ins浓缩的)”},'剂量',type2)
diabSim = SimFunction参数:可见:名字类型的单位 _____________________ ___________ _______________________ {'[ 等离子体Glu浓缩的]}{“物种”}{毫克/分升的}{'[血浆Ins浓缩的]}{“物种”}{“皮摩尔/升”}给:TargetName TargetDimension __________ _____________________ {' 剂量的}{的质量(例如,克)}
在模拟开始时,选择一份代表78克葡萄糖的单餐剂量。
singleMeal = sbioselect (m1,“名字”,'单粉');
将剂量信息转换为表格格式。
含餐= gettable(单片机);
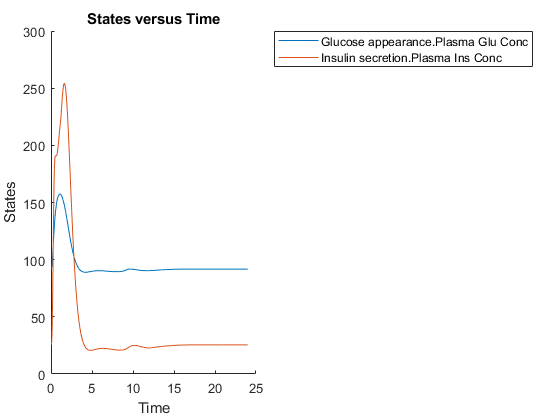
模拟正常受试者24小时的葡萄糖-胰岛素反应。
SBIOPLOT(NORMSIM([],24,含餐));
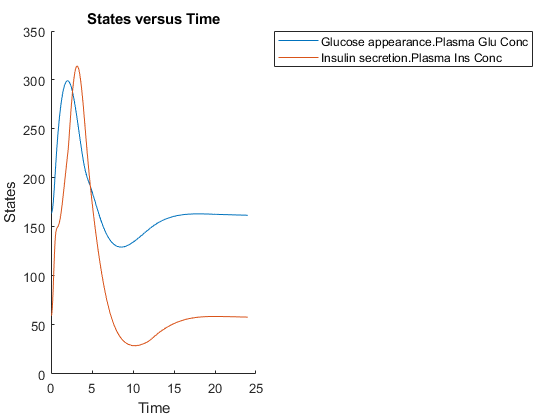
模拟糖尿病患者24小时的葡萄糖-胰岛素反应。
SBIOPLOT(贴膜([],24,含餐));
使用Variants执行扫描
假设您希望使用包含不同胰岛素损害的不同初始条件的变量数组执行参数扫描。例如,模型m1有对应于低胰岛素敏感性和高胰岛素敏感性的变异。您可以通过对SimFunction对象的单个调用来模拟这两种情况下的模型。
选择要扫描的变体。
varToScan = sbioselect (m1,“名字”,......{“低胰岛素敏感性”,'高胰岛素敏感性'});
检查每个变量中存储了哪些模型参数。
Vartoscan(1)
ans =素质变异 - 低胰岛素敏感性(非活动)contentIndex:类型:名称:属性:值:1参数Vmx值0.0235 2参数KP3值0.0045
Vartoscan(2)
ans =素质变异 - 高胰岛素敏感性(无效)contentIndex:类型:名称:属性:value:1参数Vmx值0.094 2参数kp3值0.018
这两种变体存储的交替值Vmx和kp3参数。创建SimFunction对象时,您需要将它们指定为输入参数。
创建一个simfunction对象以扫描变量。
VariantScan = CreateSimfunction(M1,{'vmx',“kp3”},......{“(等离子Glu浓缩)”,“(血浆Ins浓缩的)”},'剂量');
模拟模型并绘制结果。跑1包括低胰岛素敏感性的仿真结果跑2.对于高胰岛素敏感性。
Sbioplot(Variantscan(Vartocan,24,24次,用餐));
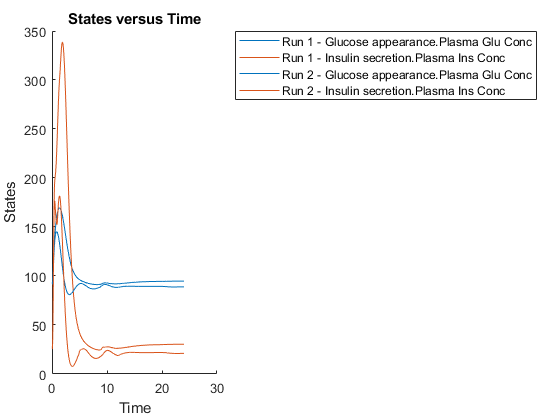
低胰岛素敏感性导致血糖浓度升高和延长。
恢复警告设置。
警告(warnSettings);
参考
D.T.吉莱斯皮(1977)。耦合化学反应的精确随机模拟。物理化学学报。81(25),2340-2361。
扩展能力
自动并行支持金宝app
通过使用并行计算工具箱™并行运行计算来加速代码。
并行运行,设置“UseParallel”到真的.
有关更多信息,请参见“UseParallel”名称-值对的论点。
另请参阅
模型对象|sbiosampleerror|sbiosampleparameters|SimBiology。场景|SIMFUNCTIP对象|SimFunctionSensitivity对象
主题
MATLAB命令
你点击一个链接对应于这个MATLAB命令:
在MATLAB命令窗口中输入它来运行命令。Web浏览器不支持MATLAB命令。金宝app

您还可以从以下列表中选择一个网站:
如何获得最佳网站性能
选择中国网站(中文或英文)以获得最佳网站性能。其他MathWorks国家站点没有针对您所在位置的访问进行优化。
