kmedoids
k-medoids集群
语法
描述
idx= kmedoids (X,k)X成k,并返回一个n1的向量idx包含每个观测值的聚类指标。行X对应点,列对应变量。默认情况下,kmedoids使用平方欧几里得距离度量和k——+ +算法用于选择初始集群中间位置。
例子
将数据分成两组
随机生成数据。
rng (“默认”);%用于再现性X = [randn(100,2)*0.75+ones(100,2);randn(100 2) * 0.55的(100 2)];图;情节(X (: 1) X (:, 2),“。”);标题(“随机生成的数据”);
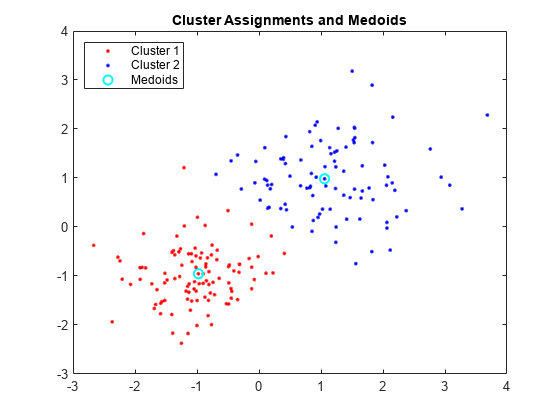
将数据分组到两个集群kmedoids.使用cityblock距离度量。
Opts = statset(“显示”,“通路”);[idx,C,sumd,d,midx,info] = kmedoids(X,2,“距离”,“cityblock”,“选项”、选择);
rep iter sum 1 1 209.856 1 2 209.856最佳距离总和= 209.856
信息是一个结构体,其中包含关于如何执行算法的信息。例如,bestReplicate字段指示用于生成最终解决方案的复制。在本例中,使用了复制数1,因为默认算法的默认复制数为1,即帕姆在这种情况下。
信息
信息=带字段的结构:算法:“pam”开始:“plus”距离:“cityblock”迭代:2 bestreplduplicate: 1
画出星团和星团中圆。
图;情节(X (idx = = 1,1) X (idx = = 1、2),“r”。,“MarkerSize”, 7)在情节(X (idx = = 2, 1), X (idx = = 2, 2),“b”。,“MarkerSize”7)情节(C (: 1), C (:, 2),“有限公司”,...“MarkerSize”7“线宽”传说,1.5)(“集群1”,《集群2》,“Medoids”,...“位置”,“西北”);标题(“集群分配和中位数”);持有从
使用k-Medoids的聚类分类数据
本例使用“Mushroom”数据集[3][4][5][6][7]来自UCI机器学习档案[7],详见http://archive.ics.uci.edu/ml/datasets/Mushroom。数据集包括22个预测因子,用于8124个不同蘑菇的观察。预测器是分类数据类型。例如,帽形是根据的特征进行分类的“b”用于钟形帽和“c”为锥形。蘑菇的颜色也有分类的特点“n”对于棕色,和“p”为粉红色。该数据集还包括每种蘑菇的可食用或有毒的分类。
由于蘑菇数据集的特征是分类的,因此不可能定义几个数据点的平均值,因此被广泛使用k-means聚类算法无法有意义地应用于该数据集。k-medoids是将数据划分为k不同的集群,通过寻找最小化数据中点和它们最近的中点之间的差异之和的中点。
一个集合的中位数是该集合中与其他集合成员的平均差异最小的成员。相似度可以定义为不允许计算平均值的许多类型的数据,允许k-medoids用于更广泛的问题比k则。
使用k-medoids,这个例子根据提供的预测器将蘑菇分为两组。然后,它探讨了这些集群和蘑菇的分类之间的关系,如可食用或有毒。
这个例子假设您已经下载了“Mushroom”数据集[3][4][5][6][7]从UCI数据库(http://archive.ics.uci.edu/ml/machine-learning-databases/mushroom/),并将其保存到当前目录中,作为一个名为agaricus-lepiota.txt的文本文件。数据中没有列标头,所以readtable使用默认变量名。
清晰的所有数据=可读数据(“agaricus-lepiota.txt”,“ReadVariableNames”、假);
展示前5个蘑菇的前几个特征。
数据(1:5,1:10)
ans = Var1 Var2 Var3 Var4 Var5 Var6 Var7 Var8 Var9 Var10 ____ ____ ____ ____ ____ ____ ____ ____ ____ _____ ' p ' ' x ' s ' ' n ' ' t ' p ' f ' c ' ' n ' k ' ' e ' x ' s ' y ' ' t ' ' ' ' f ' c ' ' b ' k ' ' e ' b ' s ' w ' ' t ' l ' f ' c ' ' b ' n ' p ' x ' y ' w ' ' t ' p ' f ' c ' ' n ' n ' ' e ' x ' s ' g ' ' f ' n ' f ' w ' ' b ' k '
提取第一列,标记数据为食用组和有毒组。然后删除该列。
Labels = data(:,1);标签= categorical(标签{:,:});Data (:,1) = [];
存储预测器(特性)的名称,在http://archive.ics.uci.edu/ml/machine-learning-databases/mushroom/agaricus-lepiota.names中有描述。
VarNames = {“cap_shape”“cap_surface”“cap_color”“淤青”“气味”...“gill_attachment”“gill_spacing”“gill_size”“gill_color”...“stalk_shape”“stalk_root”“stalk_surface_above_ring”...“stalk_surface_below_ring”“stalk_color_above_ring”...“stalk_color_below_ring”“veil_type”“veil_color”“ring_number”....“ring_type”“spore_print_color”“人口”“栖息地”};
设置变量名。
data.Properties.VariableNames = VarNames;
总共有2480个缺失值,表示为“?”.
sum (char(数据{:,:})= =“?”)
Ans = 2480
根据对数据集的检查及其描述,缺失的值只属于第11个变量(stalk_root).从表中删除列。
Data (:,11) = [];
kmedoids只接受数值数据。您需要将您拥有的类别转换为数字类型。用于定义数据不相似度的距离函数将基于分类数据的双重表示。
Cats = categorical(data{:,:});数据=双(猫);
kmedoids可以使用pdist2支持的任何距离度量来进行集群。金宝app对于本例,您将使用汉明距离对数据进行聚类,因为这是一个适用于分类数据的距离度量,如下所示。两个向量之间的汉明距离是不同向量分量的百分比。例如,考虑这两个向量。
V1 = [1 0 2 1];
V2 = [1 1 2 1];
它们在第一,三,四坐标上相等。由于4个坐标中的1个不同,这两个向量之间的汉明距离是。25。
你可以使用这个函数pdist2测量第一行和第二行数据之间的汉明距离,数值表示分类蘑菇数据。值。2857意味着蘑菇的21个特征中有6个不同。
: pdist2(数据(1),数据(2:)“汉明”)
Ans = 0.2857
在本例中,您将根据特征将蘑菇数据聚类为两个聚类,以查看聚类是否对应于可食性。的kmedoids函数保证收敛到聚类准则的局部最小值;然而,这可能不是这个问题的全局最小值。类对该问题进行几次聚类是个好主意“复制”参数。当“复制”设置为一个值,n,大于1,则运行k-medoids算法n,并返回最好的结果。
运行kmedoids要根据汉明距离将数据聚类为2个聚类,并返回3个重复的最佳结果,您可以运行以下命令。
rng (“默认”);%用于再现性[IDX, C, SUMD, D, MIDX, INFO] = kmedoids(data,2,“距离”,“汉明”,“复制”3);
让我们假设预测的第一组蘑菇是有毒的,第二组蘑菇都是可食用的。为了确定聚类结果的性能,根据已知标签,计算组1中有多少蘑菇确实有毒,组2中有多少蘑菇是可食用的。换句话说,计算假阳性、假阴性,以及真阳性和真阴性的数量。
构造一个混淆矩阵(或匹配矩阵),其中对角线元素分别表示真阳性和真阴性的数量。非对角线元素分别表示假阴性和假阳性。为方便起见,请使用confusionmat函数,计算已知标签和预测标签的混淆矩阵。中获取预测的标签信息IDX变量。IDX每个数据点包含值1和2,分别表示有毒组和可食用组。
predLabels =标签;为预测的标签初始化一个向量。predLabels(IDX==1) = category ({“p”});指定第1组是有毒的。predLabels(IDX==2) = categorical({“e”});指定第2组为可食用。confMatrix = confemat (labels,predLabels)
confMatrix = 4176 32 816 3100
在4208个食用菌中,4176个被正确预测为第2组(食用组),32个被错误预测为第1组(有毒组)。同样,在3916种有毒蘑菇中,有3100种被正确预测为第1组(有毒组),816种被错误预测为第2组(可食用组)。
给出这个混淆矩阵,计算准确率,这是真实结果(真阳性和真阴性)与整体数据的比例,以及精度,这是真实阳性结果与所有阳性结果(真阳性和假阳性)的比例。
精度= (confMatrix(1,1)+confMatrix(2,2))/(sum(sum(confMatrix)))
准确度= 0.8956
precision = confMatrix(1,1) / (confMatrix(1,1)+confMatrix(2,1))
精度= 0.8365
结果表明,将k-medoids算法应用于蘑菇的分类特征,可以得到与可食性相关的聚类。
输入参数
X- - - - - -数据
数字矩阵
数据,指定为数值矩阵。一排排的X对应于观察结果,列对应于变量。
k- - - - - -中位数
正整数
数据中的中位数,指定为正整数。
名称-值参数
的可选逗号分隔对名称,值参数。的名字参数名称和价值对应的值。的名字必须出现在引号内。您可以以任意顺序指定多个名称和值对参数Name1, Value1,…,的家.
“距离”,“欧几里得”、“复制”、3、“选项”,statset (UseParallel, 1)指定欧几里得距离,在不同起始值处的三个复制中位数,以及使用并行计算。
算法- - - - - -算法寻找中位数
“帕姆”|“小”|“克拉拉”|“大”
查找中位数的算法,指定为由逗号分隔的对组成“算法”而且“帕姆”,“小”,“克拉拉”,或“大”.的行数决定默认算法X.
的行数
X小于3000,“帕姆”为默认算法。如果行数在3000到10000之间,
“小”为默认算法。对于所有其他情况,
“大”为默认算法。
可以通过显式声明算法来覆盖默认选项。下表总结了可用的算法。
| 算法 | 描述 |
|---|---|
“帕姆” |
围绕中间体划分(PAM)是求解问题的经典算法k中描述的-medoids问题[1].在应用初始化函数来选择初始的中间体位置之后,程序执行PAM算法的交换步骤,即在中间体和非中间体之间搜索所有可能的交换,以查看中间体到集群成员距离的和是否减小。方法指定要使用的初始化函数 算法进行如下。
算法迭代构建和交换步骤,直到中间值不变,或者满足其他终止条件。 在某些情况下,该算法可以产生比其他算法更好的解,但它可能运行时间过金宝搏官方网站长。 |
“小” |
使用类似于k-means算法的算法来查找 算法进行如下。
算法重复这些步骤,直到没有进一步的更新发生或满足其他终止条件。该算法有一个可选的类似pam的在线更新阶段(由 |
“克拉拉” |
集群大型应用程序(CLARA)[1]在数据的随机子集上重复执行PAM算法。它旨在通过采样克服PAM算法带来的缩放挑战。 算法进行如下。
算法重复这些步骤,直到中位数不变,或者满足其他终止条件。 为了获得最佳性能,建议您执行多个复制。默认情况下,程序执行五次复制。每个重复样本 |
“大” |
这类似于 |
例子:“算法”、“帕姆的
OnlinePhase- - - - - -标志来执行类似pam的在线更新阶段
“上”(默认)|“关闭”
执行类似pam的在线更新阶段的标志,指定为逗号分隔的对,由“OnlinePhase”而且“上”或“关闭”.
如果是的话在,然后kmedoids的Lloyd迭代之后,对中位元执行类似pam的更新小而且大算法。在这个在线更新阶段,算法在每个聚类中选择距离medoid最远和最近的一小部分数据点。对于每个选定的点,它会重新分配整个数据集的聚类,并检查这样创建的距离之和是否小于已知的距离之和。
换句话说,交换的考虑仅限于靠近中脊和远离中脊的点。为了细化聚类,考虑了临近点。考虑远点以避免局部极小值。打开这个特性可以提高两种算法生成的解决方案的质量。金宝搏官方网站总的运行时间也会增加,但是增加的时间通常少于PAM的一次迭代。
例子:OnlinePhase,“了”
距离- - - - - -距离度量
“sqEuclidean”(默认)|“欧几里得”|特征向量|字符串标量|函数处理|……
距离度量,指定为下表中描述的距离度量的名称,或函数句柄。kmedoids最小化中间体到集群成员距离的总和。
| 价值 | 描述 |
|---|---|
“sqEuclidean” |
欧式距离的平方(默认) |
“欧几里得” |
欧氏距离 |
“seuclidean” |
标准化欧氏距离。观测值之间的每个坐标差都通过除以相应的标准偏差元素来缩放, |
“cityblock” |
城市街区距离 |
闵可夫斯基的 |
闵可夫斯基距离。指数是2。 |
“chebychev” |
切比雪夫距离(最大坐标差) |
“mahalanobis” |
的样本协方差的马氏距离 |
的余弦 |
1减去点间夹角的余弦(作为向量处理) |
“相关” |
1减去点之间的样本相关性(作为值序列处理) |
“枪兵” |
1减去观测值之间的样本斯皮尔曼秩相关(作为值序列处理) |
“汉明” |
汉明距离,也就是不同坐标的百分比 |
“jaccard” |
1减去杰卡德系数,也就是非零坐标的百分比 |
@ |
自定义距离函数句柄。距离函数有这样的形式 函数D2 = distfun(ZI,ZJ)距离计算%...
如果数据不是稀疏的,通常可以通过使用内置距离而不是函数句柄来更快地计算距离。 |
有关每个距离度量的定义,请参见距离度量.
例子:“距离”,“汉明”
选项- - - - - -控制迭代算法的选项,使拟合条件最小化
[](默认)|返回的结构数组statset
控制迭代算法以最小化拟合条件的选项,指定为由逗号分隔的对组成“选项”和返回的结构数组statset.金宝app结构数组的受支持字段指定用于控制迭代算法的选项。该表总结了支持的字段。金宝app
| 场 | 描述 |
|---|---|
显示 |
显示输出的级别。的选择是“关闭”(默认),“通路”. |
麦克斯特 |
允许的最大迭代次数。默认为One hundred.. |
UseParallel |
如果真正的,并行计算。如果并行计算工具箱™不可用,则以串行模式进行计算。默认为假,意思是串行计算。 |
UseSubstreams |
设置为真正的并行计算以可重复的方式并行计算默认为假.若要可重复计算,还必须设置流到允许子流的类型:“mlfg6331_64”或“mrg32k3a”. |
流 |
一个RandStream对象或此类对象的单元格数组。有关统计和机器学习工具箱™中这些选项和并行计算的详细信息,请参见加速统计计算或输入帮助parallelstats在命令行。 |
例子:“选项”,statset(“显示”,“关闭”)
复制- - - - - -使用新的初始聚类中间位置重复聚类的次数
正整数
使用新的初始集群中位数位置重复聚类的次数,指定为正整数。缺省值取决于算法的选择。为帕姆而且小,默认为1。为克拉拉,默认为5。为大,默认为3。
例子:“复制”,4
NumSamples- - - - - -执行时要从数据中获取的样本数克拉拉算法
40 + 2 * k(默认)|正整数
方法时从数据中提取的样本数克拉拉算法,指定为正整数。默认的样本数量计算为40 + 2 * k.
例子:“NumSamples”,160年
PercentNeighbors- - - - - -使用大型算法对数据集的百分比进行检验
0.001(默认)|0到1之间的标量值
百分比的数据集来检查使用大算法,指定为正数。
程序检查percentneighbors *大小(X, 1)medoids的邻居数。如果簇内距离和没有改善,则算法终止。
该参数的值在0到1之间,其中接近1的值往往给出高质量的解决方案,但算法需要更长的时间运行,而接近0的值往往给出低质量的解决方案,但完成得更快。金宝搏官方网站
例子:“PercentNeighbors”,0.01
开始- - - - - -选择初始簇中间位置的方法
“+”(默认)|“样本”|“集群”|矩阵
选择初始群集中间位置的方法,指定为逗号分隔的对,由“开始”而且“+”,“样本”,“集群”,或矩阵。下表总结了可用的方法。
| 方法 | 描述 |
|---|---|
“+”(默认) |
选择k观察从X根据k——+ +算法用于初始化集群中心。 |
“样本” |
选择k观察从X在随机的。 |
“集群” |
的随机子样本(10%)进行初步聚类阶段X.这个初始阶段本身就是使用样本也就是说,观测结果是随机选择的。 |
| 矩阵 | 一个自定义k——- - - - - -p起始位置的矩阵。在这种情况下,你可以进来[]为k输入参数,和kmedoids推断k从矩阵的第一维开始。您还可以提供一个3-D数组,这意味着为的值“复制”从数组的第三维。 |
例子:“开始”、“样本”
数据类型:字符|字符串|单|双
输出参数
idx- Medoid指数
数值列向量
中位数索引,作为数值列向量返回。idx有多少行X,每一行表示对应观测值的中位数赋值。
C—集群medoid位置
数字矩阵
集群中位数位置,作为数字矩阵返回。C是一个k——- - - - - -p矩阵,其中行j是集群的中位数吗j
sumd-点到中端距离的集群内和
数值列向量
点到中位数距离的集群内和,作为数值列向量返回。sumd是一个k-by1向量,其中元素j是簇内点到中间点距离的和吗j.
D-从每个点到每个中点的距离
数字矩阵
从每个点到每个中间点的距离,作为数字矩阵返回。D是一个n——- - - - - -k矩阵,其中元素(j,米)为观测距离j对medoid米.
信息-算法信息
结构体
算法信息,作为结构返回。信息包含函数在执行时使用的选项,例如k-medoid聚类算法(算法),用于选择初始聚类中间位置的方法(开始),距离度量(距离),最佳复制的迭代次数(迭代)及返回结果的复制编号(bestReplicate).
更多关于
k-medoids集群
k-medoids聚类是一种划分方法,通常用于需要对离群数据、任意距离指标或平均值或中位数没有明确定义的领域。
它类似于k-均值,这两种方法的目标都是将一组测量值或观测值划分为k子集或聚类,使子集最小化度量值与度量值的聚类中心之间的距离之和。在k-means算法,子集的中心是子集中测量值的平均值,通常称为质心。在k-medoids算法中,子集的中心是子集的一个成员,称为medoid。
的k-medoids algorithm返回数据集中的实际数据点。这允许您在数据集中不存在数据平均值的情况下使用该算法。这是两者的主要区别k-medoids和k-表示质心返回的位置k-means可能不在数据集中。因此k-medoids对于无法定义或解释平均值的分类数据的聚类非常有用。
这个函数kmedoids提供了几种迭代算法,在所有聚类中最小化每个对象到其聚类中间值的距离之和。其中一种算法叫做围绕中间点划分(PAM)[1]它分两步进行。
Build-step:每个k集群与一个潜在的中间体相关联。属性指定的技术执行此赋值
“开始”名称-值对参数。交换步骤:在每个簇中,通过检查使用该点作为中间点的簇内距离之和是否变小,将每个点作为潜在中间点进行测试。如果是,则将该点定义为新中位数。然后将每个点分配给具有最近中位数的群集。
算法迭代构建和交换步骤,直到中间值不变,或者满足其他终止条件。
您可以使用几个可选的输入参数来控制最小化的细节kmedoids,包括簇中位数的初始值和最大迭代次数。默认情况下,kmedoids使用k——+ +算法用于集群中位体初始化和平方欧几里得度规来确定距离。
参考文献
[1]考夫曼,L.和卢梭,P. J.(2009)。在数据中寻找组:聚类分析导论。新泽西州霍博肯:约翰·威利父子公司
[2] Park H-S和Jun C-H。(2009)。一种简单快速的k - meddoids聚类算法。专家系统与应用。36,3336-3341。
[3] Schlimmer, j.s(1987)。通过表征调整获得概念(技术报告87-19)。加州大学欧文分校信息与计算机科学系博士论文。
[4] Iba, W。,Wogulis,J., and Langley,P. (1988). Trading off Simplicity and Coverage in Incremental Concept Learning. In Proceedings of the 5th International Conference on Machine Learning, 73-79. Ann Arbor, Michigan: Morgan Kaufmann.
杜赫W, a.r.,和Grabczewski, K.(1996)使用反向传播网络从训练数据中提取逻辑规则。第1届软计算在线研讨会,19-30,第25-30页。
杜赫,W., Adamczak, R., Grabczewski, K.,石川,M.和上田,H.(1997)。利用约束反向传播网络提取清晰逻辑规则-两种新方法的比较。欧洲人工神经网络研讨会(ESANN'97),布鲁日,比利时16-18。
[7]巴赫,K.和利希曼,M.(2013)。UCI机器学习知识库[http://archive.ics.uci.edu/ml]。加州欧文市:加州大学信息与计算机科学学院。
扩展功能
自动平行支撑金宝app
通过使用并行计算工具箱™自动并行运行计算来加速代码。
要并行运行,请指定“选项”参数中的名称-值,并将“UseParallel”字段的选项结构为真正的使用statset.
例如:“选项”,statset (UseParallel,真的)
有关并行计算的更多信息,请参见运行MATLAB函数与自动并行支持金宝app(并行计算工具箱).
另请参阅
MATLAB命令
你点击了一个对应于这个MATLAB命令的链接:
在MATLAB命令窗口中输入该命令来运行该命令。Web浏览器不支持MATLAB命令。金宝app

您也可以从以下列表中选择一个网站:
如何获得最佳的网站性能
选择中国站点(中文或英文)以获得最佳站点性能。其他MathWorks国家站点没有针对您所在位置的访问进行优化。
