大尺寸多分辨率图像的深度学习分类
这个例子展示了如何训练一个Inception-v3深度神经网络来分类不适合存储的多分辨率完整幻灯片图像(WSIs)。
该示例演示了如何训练一个Inception-v3肿瘤分类网络,并提供了一个预训练的Inception-v3网络。如果您选择训练incepin -v3网络,强烈建议使用具有3.0或更高计算能力的NVIDIA™GPU。使用GPU需要并行计算工具箱™。
介绍
诊断乳腺癌的唯一确定方法是检查从活检或手术中收集的组织样本。通常用苏木精和伊红(H&E)染色制备样品,以增加组织结构的对比度。传统上,病理学家在显微镜下通过玻片检测肿瘤组织。诊断需要时间,因为病理学家必须在近镜下彻底检查整个切片。此外,病理学家可能不会注意到小肿瘤。深度学习方法旨在实现肿瘤组织的自动化检测,节省时间,提高小肿瘤的检出率。
肿瘤分类的深度学习方法依赖于数字化病理,将整个组织切片进行成像和数字化。产生的WSIs具有极高的分辨率。WSIs通常存储在一个多分辨率文件中,以方便图像的显示、导航和处理。
读取WSIs是一个挑战,因为图像不能作为一个整体加载到内存中,因此需要核心外的图像处理技术。图像处理工具箱™bigimage对象可以存储和处理这类大的多分辨率图像。bigimageDatastore对象可以从中准备训练面片bigimage将其输入神经网络
这个例子展示了如何训练一个深度学习网络来对非常大的多分辨率图像中的肿瘤进行分类bigimage和bigimageDatastore.该示例以热图的形式显示分类结果,描述了局部组织癌变的概率。肿瘤区域的定位使医学病理学家能够调查特定的区域,并在图像中快速识别任何大小的肿瘤。
训练数据下载
本示例使用Camelyon16挑战赛的WSIs [1].该挑战的数据包含来自两个独立来源的400个淋巴结wsi,分为270张训练图像和130张测试图像。WSIs以带有11级金字塔结构的剥离格式作为TIF文件存储。
bigimage对象警告当图像包含剥离格式,因为这种格式是缓慢的过程。为了避免在命令窗口中显示的警告,关闭而示例运行预警并在完成恢复警告状态。
warningState =警告(“关闭”,'图片:bigimage:singleStripTiff');c = onCleanup(@()警告(warningState));
训练数据集由159个正常淋巴结WSIs和111个肿瘤和健康组织淋巴结WSIs组成。通常,肿瘤组织只是健康组织的一小部分。病灶边界的地面真坐标伴随肿瘤图像。
每次培训文件的大小约为2 GB。如果您不想要下载的训练数据集或训练网络,然后直接去下载并预处理测试数据节。
创建一个目录来存储训练数据集。
trainingImageDir = fullfile (tempdir,“Camelyon16”,“培训”);如果〜存在(培训yemageager,“dir”)Mkdir(TrainingImagedir);mkdir(Fullfile(Strafticimagedir,“正常”)); mkdir(完整文件(培训图像目录),“肿瘤”)); mkdir(完整文件(培训图像目录),“lesion_annotations”));结束trainNormalDataDir = fullfile (trainingImageDir,“正常”);trainTumorDataDir = fullfile (trainingImageDir,“肿瘤”);trainTumorAnnotationDir = fullfile (trainingImageDir,“lesion_annotations”);
要下载训练数据,请到Camelyon17网站,点击第一个“CAMELYON16数据集”链接。打开“培训”目录下,然后按照下列步骤。
下载“lesion_annotations.zip”文件。将文件解压缩到由此指定的目录
trainTumorAnnotationDir多变的。打开“normal”目录。将映像下载到指定的目录
Trainnormaldatadir.多变的。打开“肿瘤”目录。将映像下载到指定的目录
trainTumorDataDir多变的。
指定训练图像的数量。需要注意的是正常组织,“normal_144.tif”的训练图像中的一个具有不能被读取的元数据bigimage对象。本示例使用剩下的158个培训文件。
numNormalFiles = 158;numTumorFiles = 111;
可视化训练数据
为了更好地理解训练数据,显示一个训练图像。因为不可能以最佳分辨率将整个图像加载到内存中,所以不能使用传统的图像显示函数,例如imshow.为了显示和处理图像数据,使用bigimage对象。
创建一个bigimage目标来自肿瘤训练图像。
tumorfilename = fullfile(traintumordatadir,“肿瘤_001.tif”);tumorImage = bigimage (tumorFileName);
检查尺寸bigimage在每个分辨率水平。级别1具有最多的像素,是最好的分辨率级别。10级具有最少量的像素,并且是最粗糙的分辨率级别。宽高比不是一致的,这表明级别并不跨越相同的世界区域。
levelSizeInfo =表((1:长度(tumorImage.LevelSizes))”,......tumorImage.LevelSizes (: 1),......tumorImage.LevelSizes (:, 2),......tumorImage.LevelSizes(:,1)。/tumorImage.LevelSizes(:,2),......'variablenames',[“分辨率水平”“图像宽度”“图像高度”“宽高比”])
levelSizeInfo=11×4表分辨率等级图像宽度图片高度纵横比________________ ___________ ____________ ____________ 1个2.2118e + 05 97792 2.2618 2 1.1059e + 05 49152 2.25 3 55296 24576 2.25 27648 4 12288 2.25 5 13824 6144 2.25 6 7168 3072 2.3333 7 3584 1536 2.3333 8 2048 1024 29 1024 512 2 10 512 512 1 11 1577 3629 0.43455
显示bigimage在粗糙的分辨率水平上使用bigimageshow函数。的句柄bigimageshow对象。您可以使用手柄来调整显示。该图像包含许多空白。该组织只占图像的一小部分。
h = bigimageshow (tumorImage,'distanceLevel',7);

放大图像的一部分,通过设置水平和垂直的空间范围,以最好的分辨率水平。图像看起来很模糊,因为这个分辨率非常粗糙。
xlim((29471、29763));ylim((117450、118110));
要查看更多详细信息,请将分辨率级别更改为更精细的级别。
h.ResolutionLevel = 1;

创建遮罩
您可以通过只处理感兴趣的区域(roi)来减少计算量。使用掩码定义roi。掩模是一种逻辑图像,其中真正的像素代表ROI。
为了进一步减小计算量,创建可完全在内存中,而不是在块逐块的基础上进行处理以粗分辨率级别掩模。如果粗分辨率级别的空间参考精细分辨率级别的空间参考相匹配,然后在粗等级对应于更精细的水平位置的位置。在这种情况下,你可以使用粗掩码在更精细的级别选择哪个块的过程。有关更多信息,请参见设置空间参考的大图和使用蒙版高效处理大图像.
指定用于创建掩码的分辨率级别。本例使用分辨率级别7,这是粗糙的,适合内存。
分辨率水平=7;
为普通图像创建蒙版
在正常图像中,ROI由健康组织组成。健康组织的颜色与背景的颜色不同,因此使用颜色阈值化划分图像并创建ROI。L * A * B *颜色空间为分割提供了最佳色彩分离。将图像转换为L * A * B *颜色空间,然后阈值A *通道以创建组织掩模。
您可以使用helper函数createMaskForNormalTissue使用颜色阈值创建蒙版。这个helper函数作为支持文件附加到示例中。金宝app
辅助函数对每个正常组织的训练图像进行以下操作:
创建一个
bigimage从TIF图像文件中获取。从图像元数据中设置所有分辨率级别的空间引用。
获得粗糙分辨率级别的图像。
将粗糙图像转换为L*a*b*颜色空间,然后提取a*通道。
通过使用Otsu的方法对图像进行阈值化,创建一个二值图像,使黑白像素之间的类内方差最小化。
创建单个分辨率
bigimage对象,并设置掩码的空间引用以匹配输入图像的空间引用。写面具
bigimage到内存中。只有bigimage对象在内存中。与逻辑掩码图像相对应的各个图像块位于临时目录中。写入目录将保留自定义空间引用,这确保普通图像及其对应的掩码图像具有相同的空间引用。
trainNormalMaskDir = fullfile (trainNormalDataDir, (“正常遮罩水平”num2str (resolutionLevel)]);Createmaskfornormaltissue(TrainNormaldatadir,Trainnormalmaskdir,DeviceLevel)
现在普通图像和遮罩都在磁盘上,创建bigimage对象来使用帮助器函数管理数据createBigImageAndMaskArrays.这个函数创建一个数组bigimage来自普通图像的对象和相应的bigimage从正常掩模图像对象。helper函数作为支持文件附加到示例中。金宝app
[bigNormalImages, bigNormalMasks] = createBigImageAndMaskArrays (trainNormalDataDir trainNormalMaskDir);
选择一个样本法向图像和蒙版。确认掩模的空间世界范围与图像的范围在最佳分辨率水平上匹配。空间世界的范围由XWorldLimits和YWorldLimits属性。
Idx = 2;BignormalImages(IDX).spatialReferencing(1)
ans = imref2d with properties: XWorldLimits: [0 97792] YWorldLimits: [0 221184] ImageSize: [221184 97792] PixelExtentInWorldX: 1 PixelExtentInWorldX: 97792 ImageExtentInWorldX: 97792 imageextentinworlddy: 221184 XIntrinsicLimits: [0.5000 9.7793e+04] YIntrinsicLimits: [0.5000 2.2118e+05]
bigNormalMasks(IDX).SpatialReferencing(1)
ANS = imref2d与属性:XWorldLimits:[0 97792] YWorldLimits:[0 221184] IMAGESIZE:[3456 1527] PixelExtentInWorldX:64.0419 PixelExtentInWorldY:64 ImageExtentInWorldX:97792 ImageExtentInWorldY:221184个XIntrinsicLimits:0.5000 1.5275e + 03] YIntrinsicLimits:[0.5000 3.4565E + 03]
验证掩码包含正确的roi和空间引用。控件显示示例图像bigimageshow函数。获取包含显示的轴。
图hBigNormal = bigimageshow(bigNormalImages(idx));hNormalAxes = hBigNormal.Parent;
在显示的顶部创建一个新的轴bigimage.在新的坐标轴中,用部分透明度显示相应的掩模图像。面罩会突出包含正常组织的区域。
hMaskAxes =轴;Hbigmask = BigimageShow(虚拟机阵列(IDX),“父”hMaskAxes,......“插值”,“最近的”,“AlphaData”, 0.5);hMaskAxes。可见=“关闭”;
用掩模的轴链接图像的轴。当您缩放和平移时,两个轴都会相同更新。
linkaxes ([hNormalAxes hMaskAxes]);

通过设置水平和垂直空间范围放大图像的一部分。遮罩与正常组织正确重叠。
xlim(80000年[45000]);ylim(165000年[130000]);

为肿瘤图像创建蒙版
在肿瘤图像中,ROI由肿瘤组织组成。肿瘤组织的颜色与健康组织的颜色相似,所以不能使用颜色分割技术。相反,通过使用伴随肿瘤图像的病灶边界的地面真值坐标来创建roi。
您可以使用helper函数createMaskForTumorTissue用投资回报来创建模板。这个helper函数作为支持文件附加到示例中。金宝app
辅助函数对每个肿瘤组织的训练图像执行以下操作:
创建一个
bigimage从TIF图像文件中获取。从图像元数据设置空间引用。
读取XML文件中相应的损伤注释,并将注释转换为多边形(
多边形对象)。对于每个图像块,使用多边形数据为对应的块创建掩码。带有肿瘤区域的图像可能包含一些正常区域。使用正常组织的注释来排除这些区域。
创建输出逻辑掩码
bigimage对象的分辨率较低。使用setBlock函数。写面具
bigimage对象指向内存中的目录。只有bigimage对象在内存中。与逻辑掩码图像相对应的各个图像块位于临时目录中。写入到目录保存自定义空间参考,这确保了肿瘤图像和它们的对应的掩模图像具有相同的空间参考。
trainTumorMaskDir = fullfile (trainTumorDataDir, (“tumor_mask_level”num2str (resolutionLevel)]);createMaskForTumorTissue (trainTumorDataDir trainTumorAnnotationDir,......trainTumorMaskDir resolutionLevel);
现在肿瘤图像和遮罩都在磁盘上,创建bigimage对象来使用帮助器函数管理数据createBigImageAndMaskArrays.这个函数创建一个数组bigimage物体从肿瘤图像和相应的阵列bigimage肿瘤掩膜图像中的物体。helper函数作为支持文件附加到示例中。金宝app
[bigTumorImages, bigTumorMasks] = createBigImageAndMaskArrays (trainTumorDataDir trainTumorMaskDir);
选择一个样本肿瘤图像和遮罩。确认掩模的空间世界范围与图像的范围在最佳分辨率水平上匹配。空间世界的范围由XWorldLimits和YWorldLimits属性。
idx = 5;bigTumorImages (idx) .SpatialReferencing (1)
ans = imref2d with properties: XWorldLimits: [0 97792] YWorldLimits: [0 219648] ImageSize: [219648 97792] PixelExtentInWorldX: 1 PixelExtentInWorldX: 97792 ImageExtentInWorldX: 97792 imageextentinworlddy: 219648 XIntrinsicLimits: [0.5000 9.77793e +04] YIntrinsicLimits: [0.5000 2.1965e+05]
bigTumorMasks (idx) .SpatialReferencing (1)
ans = imref2d with properties: XWorldLimits: [0 97792] YWorldLimits: [0 219648] ImageSize: [3432 1527] PixelExtentInWorldX: 64.0419 PixelExtentInWorldX: 97792 imageextentinworlddy: 219648 XIntrinsicLimits: [0.5000 1.5275e+03] YIntrinsicLimits: [0.5000 3.4325e+03]
验证掩码包含正确的roi和空间引用。控件显示示例图像bigimageshow函数。获取包含显示的轴。
图hBigTumor = bigimageshow(bigTumorImages(IDX));hTumorAxes = hBigTumor.Parent;
创建上所显示的大图像的顶部一个新的坐标轴。在新的坐标轴中,用部分透明度显示相应的掩模图像。面罩会突出包含正常组织的区域。
hMaskAxes =轴;hBigMask = bigimageshow (bigTumorMasks (idx),“父”hMaskAxes,......“插值”,“最近的”,“AlphaData”, 0.5);hMaskAxes。可见=“关闭”;
用掩模的轴链接图像的轴。当您缩放和平移时,两个轴都会相同更新。
Linkaxes([htumoraxes,hmaskakes]);

通过设置水平和垂直空间范围,放大图像的一部分。面罩正确地覆盖了肿瘤组织。
XLIM([45000 65000]);ylim([130000 150000]);

创建用于培训和验证的大图像数据存储
为了从训练数据的补丁bigimage对象,使用bigimageDatastore.这个数据存储读取的补丁bigimage单一分辨率级别的数据。
原始训练补丁中的颜色不平衡和类别不平衡可能会使网络产生偏差。颜色不平衡源于组织的不均匀颜色染色。类别不平衡源于数据中肿瘤和正常组织的数量不等。要纠正这些不平衡,您可以预处理和扩充数据存储。
这个例子展示了如何创建一个bigimageDatastore提取肿瘤和正常斑块来训练神经网络。该示例还展示了如何预处理和扩充数据存储以避免对网络造成偏差。
选择要读取的正常组织块位置
将正常图像和相应的遮罩随机分为两组。验证集包含两个随机选择的图像和相应的遮罩。训练集包含剩余的图像和遮罩。
normalValidationIdx = randi(numNormalFiles,[1 2]);normalTrainIdx = setdiff (1: numNormalFiles, normalValidationIdx);
与图像中特征的大小相比,修补程序的大小较小。默认情况下bigimageDatastore提取无重叠、无间隙的patch,生成大量的训练patch。您可以通过指定补丁的子集来减少训练数据量。属性指定补丁的坐标selectBlockLocations函数。方法在采样的训练补丁之间添加一个间隙BlockOffsets名称-值参数。请指定一个大于补丁大小的偏移量。在默认值0.5的基础上增加包含阈值,使网络在相对均匀的patch上训练。
patchSize = (299299 3);normalStrideFactor = 10;blsNormalData = selectBlockLocations (bigNormalImages (normalTrainIdx),......“BlockSize”patchSize (1:2),“BlockOffsets”patchSize (1:2) * normalStrideFactor,......“面具”bigNormalMasks (normalTrainIdx),“InclusionThreshold”, 0.75,“ExcludeIncompleteBlocks”,真正的);
选择要读取的验证修补程序的位置。由于验证图像较少,因此您无需在修补程序之间添加间隙。
blsNormalDataValidation = selectBlockLocations (bigNormalImages (normalValidationIdx),......“BlockSize”patchSize (1:2),......“面具”bigNormalMasks (normalValidationIdx),“InclusionThreshold”, 0.75,“ExcludeIncompleteBlocks”,真正的);
为普通映像创建数据存储
创建数据存储dsNormalData和dsNormalDataValidation从正常图像中以最佳分辨率读取图像补丁,分别用于训练和验证。属性指定补丁的坐标BlockLocations.名称-值对的论点。
dsNormalData = bigimageDatastore (bigNormalImages (normalTrainIdx),......“BlockLocationSet”, blsNormalData);dsNormalDataValidation = bigimageDatastore (bigNormalImages (normalValidationIdx),......“BlockLocationSet”,blsNormalDataValidation);
预览包含正常训练图像的数据存储中的补丁。
imagesToPreview=零([patchSize 10],“uint8”);为n=1:10 im=读取(dsNormalData);imagesToPreview(:,:,:,n)=im{1};结束图蒙太奇(想象力,“大小”(2 - 5),“BorderSize”10,'背景颜色',“k”);标题(“正常组织训练片”)

选择要读取的肿瘤组织块位置
将肿瘤图像和相应的掩模随机分为两组。验证集包含两个随机选择的图像和相应的掩模。训练集包含剩余的图像和掩模。
tumorValidationIdx = randi(numTumorFiles,[1 2]);tumorTrainIdx = setdiff (1: numTumorFiles, tumorValidationIdx);
属性指定要读取的补丁的坐标selectBlockLocations函数。肿瘤组织比正常组织更稀疏,因此可以通过指定一个比正常组织更小的块偏移来增加采样密度。注意,如果您想使用更少的训练图像进行训练,那么您可能需要通过进一步减少块偏移来增加训练集的大小。
tumorStrideFactor = 4;blsTumorData = selectBlockLocations (bigTumorImages (tumorTrainIdx),......“BlockSize”patchSize (1:2),“BlockOffsets”patchSize (1:2) * tumorStrideFactor,......“面具”bigTumorMasks (tumorTrainIdx),“InclusionThreshold”, 0.75,“ExcludeIncompleteBlocks”,真正的);
选择要读取的验证修补程序的位置。由于验证图像较少,因此您无需在修补程序之间添加间隙。
blsTumorDataValidation=选择BlockLocations(bigTumorImages(tumorValidationIdx),......“BlockSize”patchSize (1:2),......“面具”bigTumorMasks (tumorValidationIdx),“InclusionThreshold”, 0.75,“ExcludeIncompleteBlocks”,真正的);
为肿瘤图像创建数据存储
创建一个bigimageDatastore从训练的肿瘤图像和面具。的数据存储dsTumorData和dsTumorDataValidation读取来自肿瘤图像在分别用于训练和验证,最精细分辨率水平图像补丁。
dsTumorData = bigimageDatastore (bigTumorImages (tumorTrainIdx),......“BlockLocationSet”, blsTumorData);dsTumorDataValidation = bigimageDatastore (bigTumorImages (tumorValidationIdx),......“BlockLocationSet”, blsTumorDataValidation);
从包含肿瘤训练图像的数据存储预览补丁。
imagesToPreview=零([patchSize 10],“uint8”);为n=1:10im=read(dsTumorData);imagesToPreview(:,:,:,n)=im{1};结束蒙太奇(imagesToPreview,“大小”(2 - 5),“BorderSize”10,'背景颜色',“k”);标题(“肿瘤组织训练片”)

颜色归一化和增强训练数据
训练图像具有不同的颜色分布,因为数据集来自不同的来源和颜色染色组织不会得到相同的染色图像。为了避免网络的偏置,还需要进行额外的预处理。
为了防止颜色变化,本例使用标准染色归一化技术对数据进行预处理。应用染色规范和增加使用变换使用辅助功能指定的自定义预处理操作Augmentandlabel..这个函数作为支持文件附加到示例中。金宝app
的Augmentandlabel.Function执行以下操作:
正常化染色使用
正常化功能 [4]。使用Macenko的方法执行染色归一化,该方法通过使用固定矩阵的颜色反卷积分离H&E颜色通道,然后使用单独的校正混合重新创建归一化图像。该函数返回归一化图像以及H&E图像。通过使用
jitterColorHSV函数。颜色抖动通过扰动图像对比度,色调,饱和度和亮度变化各贴片的颜色。颜色抖动在HSV颜色空间中进行,以避免将RGB图像中不需要的颜色伪像。应用90度旋转以及垂直和水平反射的随机组合。随机仿射变换使网络对输入图像数据的方向不可知。
将补丁标为
“正常”或“肿瘤”.
每个图像块生成五个增强和标记补丁:stain-normalized补丁,stain-normalized补丁与颜色抖动,stain-normalized贴片颜色抖动和随机仿射变换,苏木精形象与随机仿射变换,和伊红图像随机仿射变换。
创建数据存储,用于转换正常的训练和验证图像,并将生成的补丁标记为“正常”.
dsLabelledNormalData =变换(dsNormalData,......@ (x,信息)augmentAndLabel (x,信息,“正常”),“IncludeInfo”,真正的);dsLabelledNormalDataValidation =变换(dsNormalDataValidation,......@ (x,信息)augmentAndLabel (x,信息,“正常”),“IncludeInfo”,真正的);
创建变换的肿瘤训练和验证的图像数据存储和标记生成补丁“肿瘤”.
dsLabelledTumorData =变换(dsTumorData,......@ (x,信息)augmentAndLabel (x,信息,“肿瘤”),“IncludeInfo”,真正的);dsLabelledTumorDataValidation =变换(dsTumorDataValidation,......@ (x,信息)augmentAndLabel (x,信息,“肿瘤”),“IncludeInfo”,真正的);
平衡肿瘤和正常分类
与正常组织相比,肿瘤图像中癌症组织的数量非常少。为了避免对包含大量正常组织和极少量肿瘤组织的类不平衡数据进行网络训练,需要进行额外的预处理。
为了防止类的不平衡,这个例子定义了一个自定义数据存储,称为randomSamplingDatastore以平衡的方式随机选择正常和肿瘤训练贴片。用于定义此自定义数据存储的脚本作为支持文件附加到示例中。有关详细信息,请参阅金宝app开发自定义数据存储.
创建自定义randomSamplingDatastore来自正常和肿瘤训练数据存储。随机抽样数据存储dsTrain在每次迭代时向网络提供迷你批量培训数据。
dsTrain=随机采样数据存储(dsLabelledTumorData,dsLabelledNormalData);
为了限制验证期间使用的补丁数量,本例定义了一个名为validationDatastore从每个类返回5个验证补丁。定义这个自定义数据存储的脚本作为支持文件附加到示例中。金宝app
创建自定义validationDatastore来自正常和肿瘤验证数据存储。
numValidationPatchesPerClass = 5;dsValidation = validationDatastore (dsLabelledTumorDataValidation,......dsLabelledNormalDataValidation numValidationPatchesPerClass);
设置初始化v3网络层
本例使用的是Inception-v3网络,这是一个卷积神经网络,它根据ImageNet数据库中的100多万张图像进行训练[3.].该网络有48层深度,可以将图像分类为1000个对象类别,如键盘、鼠标、铅笔和许多动物。该网络期望图像输入尺寸为299 × 299,有3个通道。
的接收v3(深度学习工具箱)函数返回一个预先训练过的初始化v3网络。启盗-v3需要深度学习工具箱™模型用于启盗-v3网络支持包。金宝app如果没有安装此支金宝app持包,则该函数将提供下载链接。
网= inceptionv3;
替换最后几层
网络的卷积层提取图像特征,最后一个可学习层和最后一个分类层用来对输入图像进行分类。这两层包含关于如何将网络提取的特征组合成类别概率、损失值和预测标签的信息。为了重新训练一个预先训练的网络来分类新的图像,将这两层替换为适应新数据集的新层。有关更多信息,请参见训练深度学习网络对新图像进行分类(深度学习工具箱).
从训练的网络中提取层图。
lgraph = layerGraph(净);
使用支持函数查找要替换的两个层的名称金宝appFindlayerStore酒店.这个函数作为支持文件附加到示例中。金宝app在《盗梦空间》v3中,这两个层被命名“预测”和“分类层预测”.
[learnableLayer, classLayer] = findLayersToReplace (lgraph)
learnableLayer = fulllyconnectedlayer with properties: Name: 'predictions' Hyperparameters InputSize: 2048 OutputSize: 1000 Learnable Parameters Weights: [1000×2048 single] Bias: [1000×1 single]显示所有属性
classLayer = ClassificationOutputLayer with properties: Name: 'ClassificationLayer_predictions' Classes: [1000×1 categorical] OutputSize: 1000 Hyperparameters LossFunction: 'crossentropyex'
这个例子的目标是在两个类之间执行二值分割,肿瘤和非肿瘤区域。为两个类创建一个新的完全连接的层。用新的图层替换原来的最终完全连接的图层。
numClasses = 2;newlearnablelayer =全连接列(numcrasses,“名字”,“预测”); lgraph=replaceLayer(lgraph,learnableLayer.Name,newLearnableLayer);
为两个类创建一个新的分类层。用新图层替换原始最终分类层。
newClassLayer = classificationLayer (“名字”,“分类层预测”);lgraph = replaceLayer (lgraph classLayer.Name newClassLayer);
指定培训选项
使用培训网络rmsprop.优化求解。该求解器自动调整学习速率和动量,以更快的收敛。属性指定其他超参数设置trainingOptions(深度学习工具箱)函数。降低MaxEpochs由于训练数据量大,使得网络更快地达到收敛。如果你有并行计算工具箱™和可用于多gpu或并行训练的硬件资源,那么通过指定ExecutionEnvironment名称-值对参数为“多gpu”或“平行”,分别。
checkpointsDir=fullfile(trainingImageDir,“检查点”);如果〜存在(CheckPointsdir,“dir”)MKDIR(CheckPointsdir);结束选择= trainingOptions (“rmsprop”,......“InitialLearnRate”1 e-5......“SquaredGradientDecayFactor”, 0.99,......“MaxEpochs”3,......“MiniBatchSize”,32,......“阴谋”,'培训 - 进步',......“CheckpointPath”checkpointsDir,......“ValidationData”dsValidation,......“ExecutionEnvironment”,“汽车”,......“洗牌”,“every-epoch”);
下载和预处理测试数据
Camelyon16测试数据集由130个WSIs组成。这些图像既有正常组织也有肿瘤组织。这个示例使用Camelyon16测试数据中的两个测试图像。每个文件的大小约为2gb。
创建一个目录来存储测试数据。
testingImageDir = fullfile (tempdir,“Camelyon16”,“测试”);如果~存在(testingImageDir“dir”mkdir (testingImageDir);mkdir (fullfile (testingImageDir,'图片')); mkdir(完整文件(testingImageDir,“lesion_annotations”));结束testdatadir = fullfile(testingimagedir,'图片');testtumorannotationdir = fullfile(testingimagedir,“lesion_annotations”);
要下载测试数据,请转到Camelyon17网站,点击第一个“CAMELYON16数据集”链接。打开“testing”目录,然后按照以下步骤操作。
下载“Descose_annotations.zip”文件。将所有文件解压缩到
testtumorannotationdir.多变的。打开“图片”目录。下载前两个文件,“test_001.tif”和“test_002.tif”。将文件移动到由指定的目录
testDataDir多变的。
指定测试映像的数量。
numtestfiles = 2;
创建测试图片面具
测试图像混合了正常和肿瘤图像。为了减少分类过程中的计算量,可以通过创建掩码来定义roi。
指定用于创建掩码的分辨率级别。本例使用分辨率级别7,这是粗糙的,适合内存。
分辨率水平=7;
为包含组织的区域创建遮罩。您可以使用helper函数createMaskForNormalTissue使用颜色阈值创建蒙版。这个helper函数作为支持文件附加到示例中。金宝app有关这个辅助函数的详细信息,请参阅为普通图像创建蒙版.
testtissuemaskdir = fullfile(testdatadir,[“test_tissuemask_level”num2str (resolutionLevel)]);createMaskForNormalTissue (testDataDir testTissueMaskDir resolutionLevel);
为包含肿瘤组织的图像创建遮罩。跳过不包含肿瘤组织的图像。您可以使用helper函数createMaskForTumorTissue创建使用ROI对象口罩。这个helper函数作为支持文件附加到示例中。金宝app有关这个辅助函数的详细信息,请参阅为肿瘤图像创建蒙版.
testTumorMaskDir = fullfile (testDataDir, (“test_tumormask_level”num2str (resolutionLevel)]);createMaskForTumorTissue (testDataDir testTumorAnnotationDir、testTumorMaskDir resolutionLevel);
列车网络的
配置培训选项和数据源之后,使用trainNetwork函数。默认情况下,本示例使用helper函数为该数据集下载预先训练过的inction -v3网络版本downloadTrainedCamelyonNet.helper函数作为支持文件附加到示例中。金宝app预先训练过的网络使您无需等待训练完成就可以运行整个示例。
注意:在NVIDIA™Titan X上的培训需要20个小时,根据你的GPU硬件,可能需要更长的时间。
doTraining = false;如果doTraining trainedNet = trainNetwork(dsTrain,lgraph,options);保存([“trainedCamelyonNet -”datestr(现在,“dd-mmm-yyyy-HH-MM-SS”)“.mat”],'trousahynet');其他的trainedCamelyonNet_url =“//www.tatmou.com/金宝appsupportfiles/vision/data/trainedCamelyonNet.mat”;netDir = fullfile (tempdir,“Camelyon16”);downloadTrainedCamelyonNet (trainedCamelyonNet_url netDir);负载(fullfile (netDir“trainedCamelyonNet.mat”));结束
下载预训练的启-V3网络Cameylon16数据。这可能需要几分钟的时间下载...已完成。
对测试数据进行分类并创建热图
强烈建议使用GPU对测试图像进行分类(需要并行计算工具箱™)。
每个测试图像有两个遮罩,一个表示正常组织,一个表示肿瘤组织。创造bigimage反对使用辅助函数来管理测试数据和口罩createBigImageAndMaskArrays.helper函数作为支持文件附加到示例中。金宝app
[bigTestImages, bigTestTissueMasks] = createBigImageAndMaskArrays (testDataDir testTissueMaskDir);[~, bigTestTumorMasks] = createBigImageAndMaskArrays (testDataDir testTumorMaskDir);
使用训练的盗梦空间,v3网络来识别测试图像肿瘤补丁,bigTestImages.使用申请函数带有helper函数指定的自定义处理管道createHeatMap.这个helper函数作为支持文件附加到示例中。金宝app若要减少所需的计算量,请指定组织掩模bigtesttissuemask.这样申请功能只处理含有组织的斑块。如果你有并行计算工具箱™和GPU,那么你可以通过指定“UseParallel”名称-值对参数为真正的.
的createHeatMapFunction对每个组织块执行以下操作:
计算肿瘤概率得分
预测(深度学习工具箱)函数。创建一个热图图像补丁,像素值等于肿瘤概率得分。
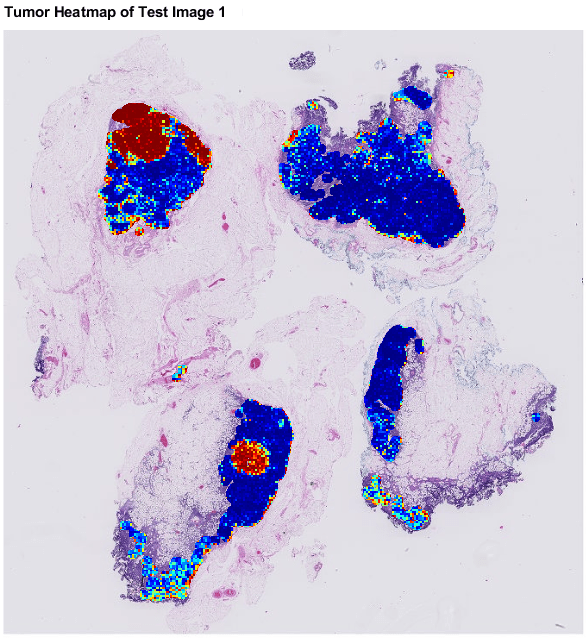
的申请函数将每个块的热图缝合为测试图像的单个热图。热图显示网络检测包含肿瘤区域的位置。
要可视化热图,将热图覆盖在原始图像上,并设置透明度“AlphaData”属性作为组织面膜。覆盖层显示肿瘤在图像中的定位程度。具有红色概率的区域具有红色像素。肿瘤概率低的区域显示为蓝色像素。
patchSize = (299299 3);为IDX = 1:numTestFiles bigTestHeatMaps(IDX)=申请(bigTestImages(IDX),1,@(X)createHeatMap(X,trainedNet),......“面具”bigTestTissueMasks (idx),'inclusionthreshold'0,......“块大小”patchSize (1:2),“UseParallel”、假);图hBigTest = bigimageshow(bigTestImages(idx));hTestAxes = hBigTest.Parent;hTestAxes。可见=“关闭”; hMaskAxes=轴;hBigMask=bigimageshow(BigTestHeatmap(idx),“父”hMaskAxes,......“插值”,“最近的”,“alphadata”,bigTestTissueMasks(idx));彩色地图(jet(255));hMaskAxes.Visible=“关闭”;linkaxes ([hTestAxes hMaskAxes]);标题([“测试图像的肿瘤热图”,num2str(idx)])结束

参考
[1] Ehteshami B.B.,等人,“检测乳腺癌妇女淋巴结转移的深度学习算法的诊断评估。”美国医学协会杂志.Vol. 318, No. 22, 2017, pp. 2199-2210。doi: 10.1001 / jama.2017.14585
Szegedy, C., V. Vanhoucke, S. Ioffe, J. Shlens, Z. Wojna。“重新思考计算机视觉的Inception架构。”在计算机视觉与模式识别会议论文集,2818-2826。拉斯维加斯,NV:IEEE,2016。
[3] ImageNet。http://www.image-net.org.
等。一种用于定量分析的组织学切片标准化方法在2009 IEEE生物医学成像国际研讨会:从纳米到宏观, 1107 - 1110。波士顿,MA: IEEE, 2009。
[5] xml2struct//www.tatmou.com/matlabcentral/fileexchange/28518-xml2struct.
另请参阅
bigimage|bigimageDatastore|bigimageshow|变换|trainingOptions(深度学习工具箱)|trainNetwork(深度学习工具箱)
相关的话题
- 用于深度学习的数据存储(深度学习工具箱)
- 深度学习层列表(深度学习工具箱)

